

Segment Solubilizing Strategy in Protein Chemical Synthesis
Received date: 2023-03-27
Revised date: 2023-07-10
Online published: 2023-08-07
Supported by
National Natural Science Foundation of China(22277073)
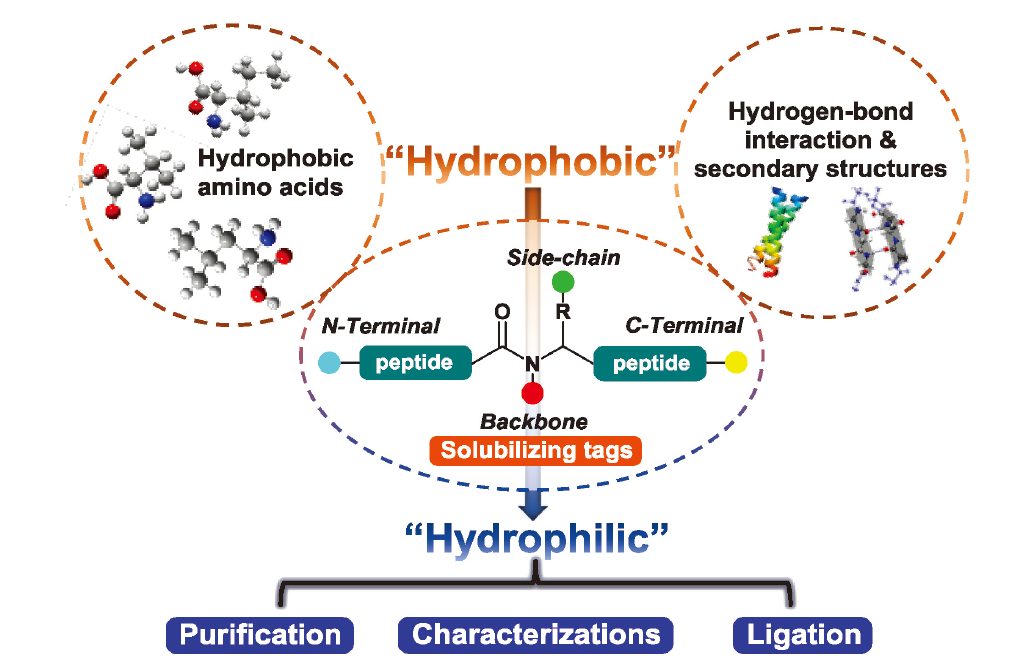
Proteins play critical roles in various biological processes and biomedical researches. A significant task of such biochemical studies is to obtain protein samples of high homogeneity with respect to their atomic compositions. Chemical protein synthesis offers a much more robust and effective strategy over recombinant expression technology for accessing proteins that are precisely modified or even artificially designed. However, some important proteins that can be used as drug targets (such as human interleukin-2, K+ channel protein Kir5.1, etc.) suffer from limited solubility of peptide segments during the journey of protein synthesis. Such hydrophobic peptides pose difficulty for subsequent purification, characterization, chemical ligation and other operations. The main factors for these problems may be that the peptide segments are prone to self-assemble into secondary structures through hydrophobic interactions, hydrogen bond or other interaction modes, thus reducing the solubility. Addition of solubilizing tags is recognized as one of the effective methods to overcome such obstacles. In this review, strategies of attaching solubilizing tags to the main chain, side chain and backbone of peptides are introduced. Membrane protein FCER1G, co-chaperone protein GroES and other proteins are selected as examples to describe the applications of the solubilizing tags. Moreover, the future of solubilizing tags strategy is discussed and prospected.
1 Introduction
2 Main chain solubilizing tags
2.1 C-terminal solubilizing tags
2.2 N-terminal solubilizing tags
3 Side chain solubilizing tags
3.1 Cysteine (Cys) side chain solubilizing tags
3.2 Lysine (Lys) side chain solubilizing tags
3.3 Asparagine (Asn) /Glutamine (Gln) side chain solubilizing tags
4 Backbone modifications as solubilizing tags
4.1 Irreversible backbone modification
4.2 Removable backbone modification
5 Conclusion and outlook
Deng Xiangyu , Zhang Baochang , Qu Qian . Segment Solubilizing Strategy in Protein Chemical Synthesis[J]. Progress in Chemistry, 2023 , 35(11) : 1579 -1594 . DOI: 10.7536/PC230325
| [1] |
Kulkarni S S, Sayers J, Premdjee B, Payne R J. Nat. Rev. Chem., 2018, 2(4): 0122.
|
| [2] |
Lin H N, Caroll K S. Chem. Rev., 2018, 118(3): 887.
|
| [3] |
Ai H S, Sun M S, Liu A J, Sun Z X, Liu T T, Cao L, Liang L J, Qu Q, Li Z C, Deng Z H, Tong Z B, Chu G C, Tian X L, Deng H T, Zhao S W, Li J B, Lou Z Y, Liu L. Nat. Chem. Biol., 2022, 18(9): 972.
|
| [4] |
Yu Y Y, Zheng Q Y, Erramilli S K, Pan M, Park S, Xie Y, Li J X, Fei J Y, Kossiakoff A A, Liu L, Zhao M L. Nat. Chem. Biol., 2021, 17(8): 896.
|
| [5] |
Pan M, Zheng Q Y, Wang T, Liang L J, Mao J X, Zuo C, Ding R C, Ai H S, Xie Y, Si D, Yu Y Y, Liu L, Zhao M L. Nature, 2021, 600(7888): 334.
|
| [6] |
Liang L J, Chu G C, Qu Q, Zuo C, Mao J X, Zheng Q Y, Chen J N, Meng X B, Jing Y, Deng H T, Li Y M, Liu L. Angew. Chem. Int. Ed., 2021, 60(31): 17171.
|
| [7] |
Pan M, Zheng Q Y, Ding S, Zhang L J, Qu Q, Wang T, Hong D N, Ren Y J, Liang L J, Chen C L, Mei Z Q, Liu L. Angew. Chem. Int. Ed., 2019, 58(9): 2627.
|
| [8] |
Zuo C, Shi W W, Chen X X, Glatz M, Riedl B, Flamme I, Pook E, Wang J W, Fang G M, Bierer D, Liu L. Sci. China Chem., 2019, 62(10): 1371.
|
| [9] |
Muttenthaler M, King G F, Adams D J, Alewood P F. Nat. Rev. Drug Discov., 2021, 20(4): 309.
|
| [10] |
Christou G A, Katsiki N, Blundell J, Fruhbeck G, Kiortsis D N. Obes. Rev., 2019, 20(6): 805.
|
| [11] |
Bassotti G, Usai-Satta P, Bellini M. Expert Opin. Pharmacother., 2018, 19(11): 1261.
|
| [12] |
Scheen A J. Expert. Opin. Biol. Ther., 2017, 17: 485.
|
| [13] |
Hamano N, Komaba H, Fukagawa M. Expert Opin. Pharmacother., 2017, 18(5): 529.
|
| [14] |
Chang R, Chen J L, Zhang G Y, Li Y, Duan H Z, Luo S Z, Chen Y X. J. Am. Chem. Soc., 2022, 144(27): 12147.
|
| [15] |
Sun J, Xiao L L, Li B, Zhao K L, Wang Z L, Zhou Y, Ma C, Li J J, Zhang H J, Herrmann A, Liu K. Angew. Chem. Int. Ed., 2021, 60(44): 23687.
|
| [16] |
Ma C, Dong J J, Viviani M, Tulini I, Pontillo N, Maity S, Zhou Y, Roos W H, Liu K, Herrmann A, Portale G. Sci. Adv., 2020, 6(29): eabc0810.
|
| [17] |
Li J J, Li B, Sun J, Ma C, Wan S K, Li Y X, Göstl R, Herrmann A, Liu K, Zhang H J. Adv. Mater., 2020, 32(17): 2000964.
|
| [18] |
Wu D D, Sinha N, Lee J, Sutherland B P, Halaszynski N I, Tian Y, Caplan J, Zhang H V, Saven J G, Kloxin C J, Pochan D J. Nature, 2019, 574(7780): 658.
|
| [19] |
Conibear A C, Watson E E, Payne R J, Becker C F W. Chem. Soc. Rev., 2018, 47(24): 9046.
|
| [20] |
Gao S, Pan M, Zheng Y, Huang Y C, Zheng Q Y, Sun D M, Lu L N, Tan X D, Tan X L, Lan H, Wang J X, Wang T, Wang J W, Liu L. J. Am. Chem. Soc., 2016, 138(43): 14497.
|
| [21] |
Pan M, Gao S, Zheng Y, Tan X D, Lan H, Tan X L, Sun D M, Lu L N, Wang T, Zheng Q Y, Huang Y C, Wang J W, Liu L. J. Am. Chem. Soc., 2016, 138(23): 7429.
|
| [22] |
Wang Z M, Xu W L, Liu L, Zhu T F. Nat. Chem., 2016, 8(7): 698.
|
| [23] |
Sun H, Brik A. Acc. Chem. Res., 2019, 52(12): 3361.
|
| [24] |
Merrifield R B. J. Am. Chem. Soc., 1963, 85(14): 2149.
|
| [25] |
Hartrampf N, Saebi A, Poskus M, Gates Z P, Callahan A J, Cowfer A E, Hanna S, Antilla S, Schissel C K, Quartararo A J, Ye X, Mijalis A J, Simon M D, Loas A, Liu S, Jessen C, Nielsen T E, Pentelute B L. Science, 2020, 368(6494): 980.
|
| [26] |
Sato K, Farquhar C E, Rodriguez J, Pentelute B L. J. Am. Chem. Soc., 2023, 145(24): 12992.
|
| [27] |
Dawson P E, Muir T W, Clark-Lewis I, Kent S B H. Science, 1994, 266(5186): 776.
|
| [28] |
Li Y M, Li Y T, Pan M, Kong X Q, Huang Y C, Hong Z Y, Liu L. Angew. Chem. Int. Ed., 2014, 53(8): 2198.
|
| [29] |
Zheng J S, Tang S, Qi Y K, Wang Z P, Liu L. Nat. Protoc., 2013, 8(12): 2483.
|
| [30] |
Fang G M, Li Y M, Shen F, Huang Y C, Li J B, Lin Y, Cui H K, Liu L. Angew. Chem. Int. Ed., 2011, 50(33): 7645.
|
| [31] |
Liu H, Li X C. Acc. Chem. Res., 2018, 51(7): 1643.
|
| [32] |
Zhang Y F, Xu C, Lam H Y, Lee C L, Li X C. Proc. Natl. Acad. Sci. U. S. A., 2013, 110(17): 6657.
|
| [33] |
Mitchell N J, Malins L R, Liu X Y, Thompson R E, Chan B, Radom L, Payne R J. J. Am. Chem. Soc., 2015, 137(44): 14011.
|
| [34] |
Tan Y, Li J S, Jin K, Liu J M, Chen Z Y, Yang J, Li X C. Angew. Chem. Int. Ed., 2020, 59(31): 12741.
|
| [35] |
Bode J W. Acc. Chem. Res., 2017, 50(9): 2104.
|
| [36] |
Pusterla I, Bode J W. Nat. Chem., 2015, 7(8): 668.
|
| [37] |
Zuo C, Zhang B C, Yan B J, Zheng J S. Org. Biomol. Chem., 2019, 17(4): 727.
|
| [38] |
Tang S, Si Y Y, Wang Z P, Mei K R, Chen X, Cheng J Y, Zheng J S, Liu L. Angew. Chem. Int. Ed., 2015, 54(19): 5713.
|
| [39] |
Fang G M, Wang J X, Liu L. Angew. Chem. Int. Ed., 2012, 51(41): 10347.
|
| [40] |
Pech A, Achenbach J, Jahnz M, Schülzchen S, Jarosch F, Bordusa F, Klussmann S. Nucleic Acids Res, 2017, 45(7): 3997.
|
| [41] |
Xu W L, Jiang W J, Wang J X, Yu L P, Chen J, Liu X Y, Liu L, Zhu T F. Cell Discov., 2017, 3: 17008.
|
| [42] |
Weinstock M T, Jacobsen M T, Kay M S. Proc. Natl. Acad. Sci. U. S. A., 2014, 111(32): 11679.
|
| [43] |
Kumar K S A, Bavikar S N, Spasser L, Moyal T, Ohayon S, Brik A. Angew. Chem. Int. Ed., 2011, 50(27): 6137.
|
| [44] |
Shi W W, Shi C W, Wang T Y, Li Y L, Zhou Y K, Zhang X H, Bierer D, Zheng J S, Liu L. J. Am. Chem. Soc., 2022, 144(1): 349.
|
| [45] |
Li Y L, Heng J, Sun D M, Zhang B C, Zhang X, Zheng Y P, Shi W W, Wang T Y, Li J Y, Sun X O, Liu X Y, Zheng J S, Kobilka B K, Liu L. J. Am. Chem. Soc., 2021, 143(42): 17566.
|
| [46] |
Wang J X, Fang G M, He Y, Qu D L, Yu M, Hong Z Y, Liu L. Angew. Chem. Int. Ed., 2015, 54(7): 2194.
|
| [47] |
Wu H X, Wei T Y, Ngai W L, Zhou H Y, Li X C. J. Am. Chem. Soc., 2022, 144(32): 14748.
|
| [48] |
Hojo H, Takei T, Asahina Y, Okumura N, Takao T, So M, Suetake I, Sato T, Kawamoto A, Hirabayashi Y. Angew. Chem. Int. Ed., 2021, 60(25): 13900.
|
| [49] |
Zhou X M, Zuo C, Li W Q, Shi W W, Zhou X W, Wang H F, Chen S M, Du J F, Chen G Y, Zhai W J, Zhao W S, Wu Y H, Qi Y M, Liu L, Gao Y F. Angew. Chem. Int. Ed., 2020, 59(35): 15114.
|
| [50] |
Zuo C, Tang S, Zheng J S. J. Pept. Sci., 2015, 21: 540.
|
| [51] |
Shen F, Huang Y C, Tang S, Chen Y X, Liu L. Isr. J. Chem., 2011, 51(8/9): 940.
|
| [52] |
Venkatakrishnan A J, Deupi X, Lebon G, Tate C G, Schertler G F, Babu M M. Nature, 2013, 494(7436): 185.
|
| [53] |
Gouaux E, MacKinnon R. Science, 2005, 310(5753): 1461.
|
| [54] |
Bill R M, Henderson P J F, Iwata S, Kunji E R S, Michel H, Neutze R, Newstead S, Poolman B, Tate C G, Vogel H. Nat. Biotechnol., 2011, 29(4): 335.
|
| [55] |
Yıldırım M A, Goh K I, Cusick M E, Barabási A L, Vidal M. Nat. Biotechnol., 2007, 25(10): 1119.
|
| [56] |
Zheng J S, Yu M, Qi Y K, Tang S, Shen F, Wang Z P, Xiao L, Zhang L H, Tian C L, Liu L. J. Am. Chem. Soc., 2014, 136(9): 3695.
|
| [57] |
Rohrbacher F, Zwicky A, Bode J W. Chem. Sci., 2017, 8(5): 4051.
|
| [58] |
Shen F, Tang S, Liu L. Sci. China Chem., 2011, 54(1): 110.
|
| [59] |
Valiyaveetil F I, MacKinnon R, Muir T W. J. Am. Chem. Soc., 2002, 124(31): 9113.
|
| [60] |
Tailhades J, Patil N A, Hossain M A, Wade J D. J. Pept. Sci., 2015, 21(3): 139.
|
| [61] |
Liu F, Luo E Y, Flora D B, Mezo A R. Angew. Chem. Int. Ed., 2014, 53(15): 3983.
|
| [62] |
Sohma Y, Chiyomori Y, Kimura M, Fukao F, Taniguchi A, Hayashi Y, Kimura T, Kiso Y. Bioorg. Med. Chem., 2005, 13(22): 6167.
|
| [63] |
Zhang B C, Li Y L, Shi W W, Wang T Y, Zhang F, Liu L. Chem. Res. Chin. Univ., 2020, 36(5): 733.
|
| [64] |
Chang H N, Liu B Y, Qi Y K, Zhou Y, Chen Y P, Pan K M, Li W W, Zhou X M, Ma W W, Fu C Y, Qi Y M, Liu L, Gao Y F. Angew. Chem. Int. Ed., 2015, 54(40): 11760.
|
| [65] |
Lahiri S, Brehs M, Olschewski D, Becker C F W. Angew. Chem. Int. Ed., 2011, 50(17): 3988.
|
| [66] |
Bianchi E, Ingenito R, Simon R J, Pessi A. J. Am. Chem. Soc., 1999, 121(33): 7698.
|
| [67] |
Johnson E C B, Kent S B H. Tetrahedron Lett., 2007, 48(10): 1795.
|
| [68] |
Loomis C R, Walsh J P, Bell R M. J. Biol. Chem., 1985, 260(7): 4091.
|
| [69] |
Sohma Y, Pentelute B, Whittaker J, Hua Q X, Whittaker L, Weiss M, Kent S B H. Angew. Chem. Int. Ed., 2008, 47(6): 1102.
|
| [70] |
Johnson E C B, Malito E, Shen Y Q, Rich D, Tang W J, Kent S B H. J. Am. Chem. Soc., 2007, 129(37): 11480.
|
| [71] |
Paradís-Bas M, Tulla-Puche J, Albericio F. Org. Lett., 2015, 17(2): 294.
|
| [72] |
Harris P W R, Brimble M A. Biopolymers, 2010, 94(4): 542.
|
| [73] |
Englebretsen D R, Robillard G T. Tetrahedron, 1999, 55(21): 6623.
|
| [74] |
Bello C, Farbiarz K, Möller J F, Becker C F W, Schwientek T. Chem. Sci., 2014, 5(4): 1634.
|
| [75] |
Englebretsen D R, Choma C T, Robillard G T. Tetrahedron Lett., 1998, 39(27): 4929.
|
| [76] |
Englebretsen D R, Choma C T, Robillard G T. Tetrahedron Lett., 1998, 39(27): 2417.
|
| [77] |
Englebretsen D R, Alewood P F. Tetrahedron Lett., 1996, 37(46): 8431.
|
| [78] |
Maity S K, Mann G, Jbara M, Laps S, Kamnesky G, Brik A. Org. Lett., 2016, 18(12): 3026.
|
| [79] |
McGinty R K, Tan S. Chem. Rev., 2015, 115(6): 2255.
|
| [80] |
Li J B, Li Y Y, He Q Q, Li Y M, Li H T, Liu L. Org. Biomol. Chem., 2014, 12(29): 5435.
|
| [81] |
Tsuda S, Mochizuki M, Ishiba H, Yoshizawa-Kumagaye K, Nishio H, Oishi S, Yoshiya T. Angew. Chem. Int. Ed., 2018, 57(8): 2105.
|
| [82] |
Birnbaum F, Nassal M. J. Virol., 1990, 64(7): 3319.
|
| [83] |
Tsuda S, Masuda S, Yoshiya T. ChemBioChem, 2019, 20(16): 2063.
|
| [84] |
Pomroy N C, Deber C M. Tetrahedron, 1999, 275(2): 224.
|
| [85] |
Liu J M, Wei T Y, Tan Y, Liu H, Li X C. Chem. Sci., 2022, 13(5): 1367.
|
| [86] |
Cao W, Rosen D B, Ito T, Bover L, Bao M S, Watanabe G, Yao Z B, Zhang L, Lanier L L, Liu Y J. J. Exp. Med., 2006, 203(6): 1399.
|
| [87] |
Agouridas V, El Mahdi O, Cargoët M, Melnyk O. Bioorg. Med. Chem., 2017, 25(18): 4938.
|
| [88] |
Hiberg-Nielsen R, Westh P, Arleth L. Biophys. J., 2009, 96(1): 153.
|
| [89] |
Tan Z P, Shang S Y, Danishefsky S J. Proc. Natl. Acad. Sci. U. S. A., 2011, 108(11): 4297.
|
| [90] |
Thevis M, Schanzer W. Mini Rev. Med. Chem., 2007, 7(5): 533.
|
| [91] |
Asahina Y, Komiya S, Ohagi A, Fujimoto R, Tamagaki H, Nakagawa K, Sato T, Akira S, Takao T, Ishii A, Nakahara Y, Hojo H. Angew. Chem. Int. Ed., 2015, 54(28): 8226.
|
| [92] |
Taniguchi T, Matsui H, Fujita T, Takaoka C, Kashima N, Yoshimoto R, Hamuro J. Nature, 1983, 302(5906): 305.
|
| [93] |
Chhabra S R, Hothi B, Evans D J, White P D, Bycroft B W, Chan W C. Tetrahedron Lett., 1998, 39(12): 1603.
|
| [94] |
Jacobsen M T, Petersen M E, Ye X, Galibert M, Lorimer G H, Aucagne V, Kay M S. J. Am. Chem. Soc., 2016, 138(36): 11775.
|
| [95] |
Ellis R J, Hemmingsen S M. Trends Biochem. Sci., 1989, 14(8): 339.
|
| [96] |
Fulcher J M, Petersen M E, Giesler R J, Cruz Z S, Eckert D M, Francis J N, Kawamoto E M, Jacobsen M T, Kay M S. Org. Biomol. Chem., 2019, 17(48): 10237.
|
| [97] |
Tsuda S, Nishio H, Yoshiya T. Chem. Commun., 2018, 54(64): 8861.
|
| [98] |
Aggeli A, Bell M, Boden N, Keen J N, Knowles P F, McLeish T C B, Pitkeathly M, Radford S E. Nature, 1997, 386(6622): 259.
|
| [99] |
Bosques C J, Imperiali B. J. Am. Chem. Soc., 2003, 125(25): 7530.
|
| [100] |
Huang Y C, Li Y M, Chen Y, Pan M, Li Y T, Yu L, Guo Q X, Liu L. Angew. Chem. Int. Ed., 2013, 52(18): 4858.
|
| [101] |
Tanida I, Ueno T, Kominami E. Methods Mol. Biol., 2008, 445: 77.
|
| [102] |
Johnson E C B, Kent S B H. J. Am. Chem. Soc., 2006, 128(22): 7140.
|
| [103] |
Li J B, Tang S, Zheng J S, Tian C L, Liu L. Acc. Chem. Res., 2017, 50(5): 1143.
|
| [104] |
Johnson T, Quibell M, Owen D, Sheppard R C. J. Chem. Soc., Chem. Commun., 1993(4): 369.
|
| [105] |
Weygand F, Steglich W, Bjarnason J, Akhtar R, Khan N M. Tetrahedron Lett., 1966, 7(29): 3483.
|
| [106] |
Zheng J S, He Y, Zuo C, Cai X Y, Tang S, Wang Z A, Zhang L H, Tian C L, Liu L. J. Am. Chem. Soc., 2016, 138(10): 3553.
|
| [107] |
Sakai A, St Claire M, Faulk K, Govindarajan S, Emerson S U, Purcell R H, Bukh J. Proc. Natl. Acad. Sci. U. S. A., 2003, 100(20): 11646.
|
| [108] |
Gan S W, Surya W, Vararattanavech A, Torres J. PLoS One, 2014, 9(1): e78494.
|
| [109] |
Tang S, Zuo C, Huang D L, Cai X Y, Zhang L H, Tian C L, Zheng J S, Liu L. Nat. Protoc., 2017, 12(12): 2554.
|
| [110] |
Zhang B C, Deng Q, Zuo C, Yan B J, Zuo C, Cao X X, Zhu T F, Zheng J S, Liu L. Angew. Chem. Int. Ed., 2019, 58(35): 12231.
|
| [111] |
Chemuru S, Kodali R, Wetzel R. Pept. Sci., 2014, 102(2): 206.
|
| [112] |
Zhang B C, Zheng Y P, Chu G C, Deng X Y, Wang T Y, Shi W W, Zhou Y K, Tang S, Zheng J S, Liu L. Angew. Chem. Int. Ed., 2023, 62(33): e202306270.
|
| [113] |
Ollivier N, SÉnÉchal M, Desmet R, Snella B, Agouridas V, Melnyk O. Nat. Commun., 2022, 13: 6667.
|
| [114] |
Chisholm T S, Kulkarni S S, Hossain K R, Cornelius F, Clarke R J, Payne R J. J. Am. Chem. Soc., 2020, 142(2): 1090.
|
| [115] |
Jin S J, Brea R J, Rudd A K, Moon S P, Pratt M R, Devaraj N K. Nat. Commun., 2020, 11: 2793.
|
/
| 〈 |
|
〉 |