Contents
1 引言
2 CRISPR-Cas的核酸分析检测技术
2.1 Cas9的核酸检测平台
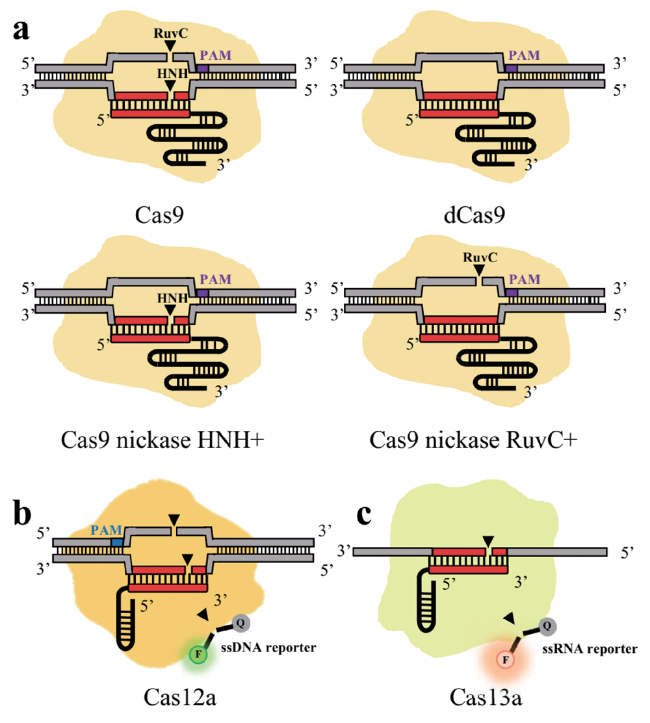
图1 CRISPR-Cas核酸酶活性概述:(a)在gRNA引导下,Cas9蛋白的RuvC和HNH核酸酶结构域可以靶向剪切DNA双链。在Cas9基础上对核酸酶结构域进行突变,产生具有切口酶活性的Cas9 nickase HNH+和Cas9 nickase RuvC+,以及没有剪切活性的dCas9。(b)Cas12a识别剪切特异序列的ssDNA或dsDNA,并激活非特异性的DNA酶活性。(c)Cas13a识别剪切特异序列的ssRNA,并激活非特异性的RNA酶活性Fig. 1 The nuclease activity of CRISPR-Cas protein: (a) Under the guidance of gRNA, the nuclease domains of Cas9 protein, RuvC and HNH, would target DNA double strands. Mutating of the nuclease domain on the basis of Cas9 can produce Cas9 nickase HNH+ and Cas9 nickase RuvC+, and deactivated dCas9. (b) Cas12a recognizes and cleaves ssDNA or dsDNA with specific sequences and activates non-specific DNase activity. (C) Cas13a recognizes and cleaves ssRNA with specific sequences and activates non-specific RNase activity |
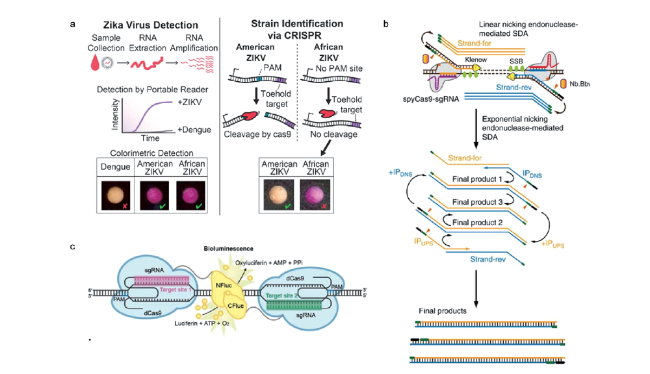
图2 基于CRISPR-Cas9的核酸检测技术:(a)在纸基试纸上利用Cas9的序列识别能力实现ZIKV病毒检测分型[37];(b)CRISPR-Cas9引发的切口酶介导的链置换扩增法(CRISDA)检测人和转基因大豆基因[40];(c)用一对荧光素酶修饰的dCas9对病原菌特征核酸序列检测[42]Fig. 2 CRISPR/Cas9 based nucleic acid detection: (a) ZIKV virus detection and genotyping on paper test strips[37]; (b) detection of human and transgenic soybean genes using CRISPR-Cas9-triggered nicking endonuclease mediated strand displacement amplification method (CRISDA)[40]; (c) detection of characteristic nucleic acid sequences of pathogenic bacteria with a pair of luciferase-modified dCas9[42] |
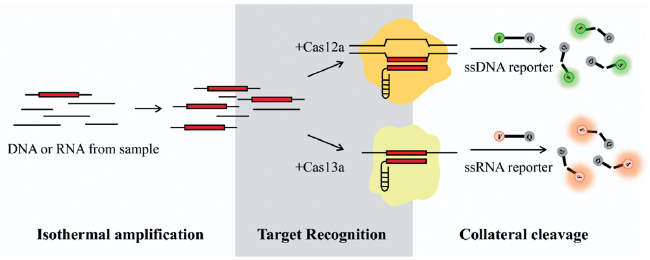
2.2 Cas13a的核酸检测平台
2.3 Cas12a的核酸检测平台
2.4 CRISPR-Cas9核酸检测的优点和不足
3 CRISPR-Cas的其他生物传感器
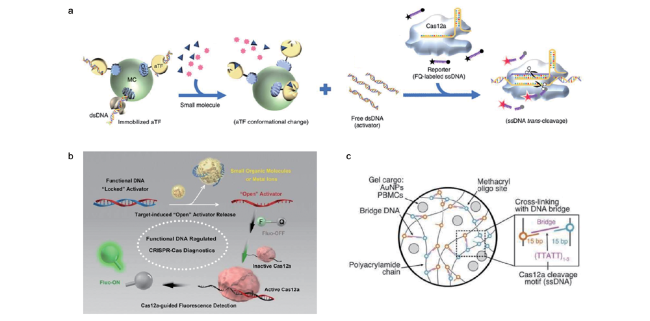
3.1 CRISPR的小分子分析方法
3.2 CRISPR智能材料的构建
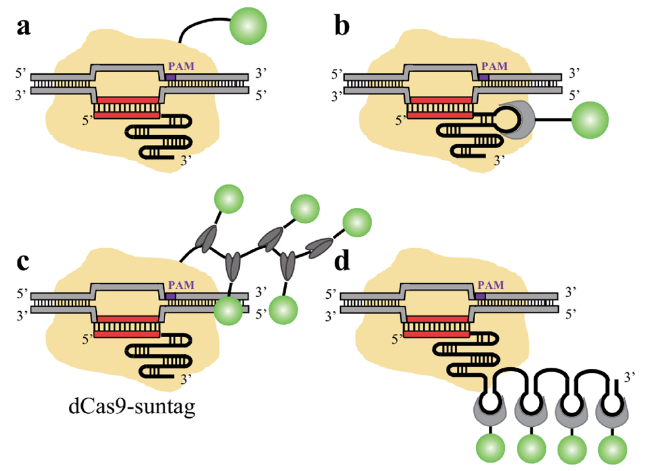
4 CRISPR的基因成像技术
图5 CRISPR成像技术原理:(a)dCas9蛋白上修饰荧光蛋白;(b)gRNA骨架中插入适配体序列,由噬菌体衣壳修饰荧光蛋白,适配体序列和噬菌体衣壳相结合;(c)在dCas9上修饰重复的抗原表位决定簇,将荧光蛋白连接在抗体上,抗体和抗原表位决定簇相结合;(d)gRNA骨架上可以修饰重复的适配体序列,招募多个噬菌体衣壳修饰的荧光蛋白Fig. 5 Illustration of CRISPR imaging technology: (a) dCas9 protein was fused with fluorescent protein; (b) The aptamer sequence was inserted into gRNA backbone and bound to a fluorescent protein fused to the phage capsid. (c) Repeated epitope determinants were fused on dCas9 and bound to antibodies fused to fluorescent proteins; (d) Repeated aptamers were inserted to the gRNA backbone to recruit multiple fluorescent proteins fused to the phage capsid |