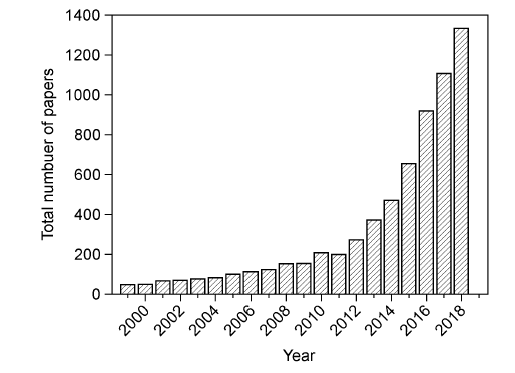
1 引言
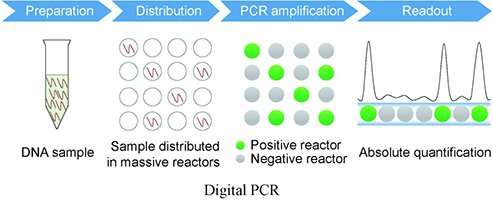
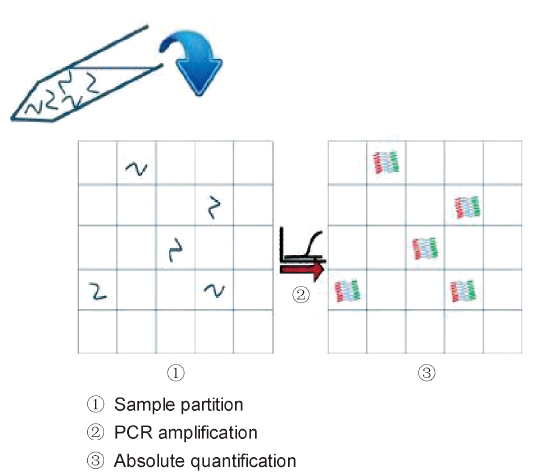
2 数字PCR技术的原理
3 数字PCR技术的发展及分类
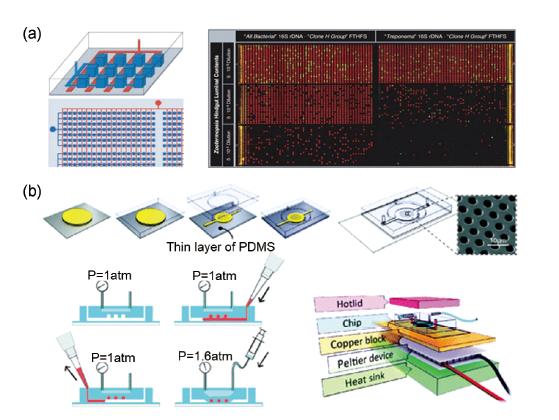
3.1 基于微反应腔室的数字PCR系统
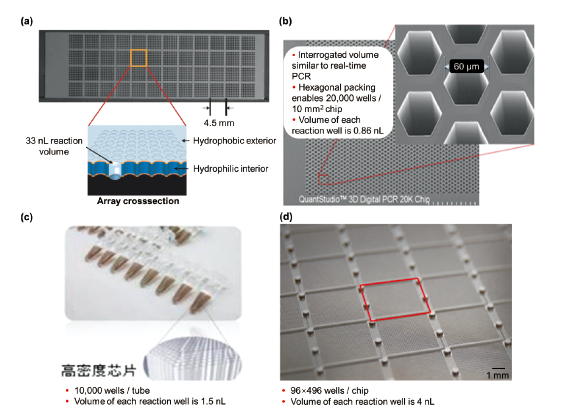
图3 基于通孔阵列的数字PCR芯片(a)不锈钢数字PCR芯片[14];(b)QuantstudioTM 3D数字PCR芯片[15];(c)ClarityTM数字PCR芯片[16];(d)Constellation数字PCR芯片[17] Fig. 3 Microhole array-based digital PCR chip (a) stainless steel digital PCR chip[14];(b) QuantstudioTM 3D digital PCR chip[15];(c) ClarityTM digital PCR chip[16];(d) Constellation digital PCR chip[17] |
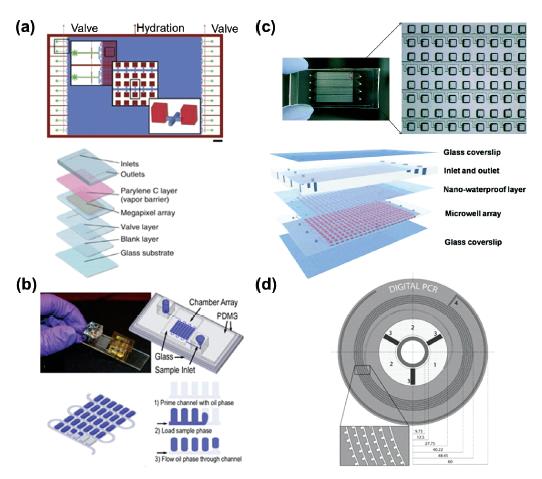
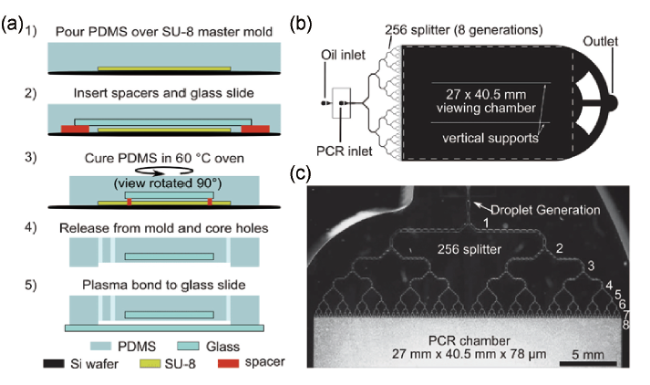
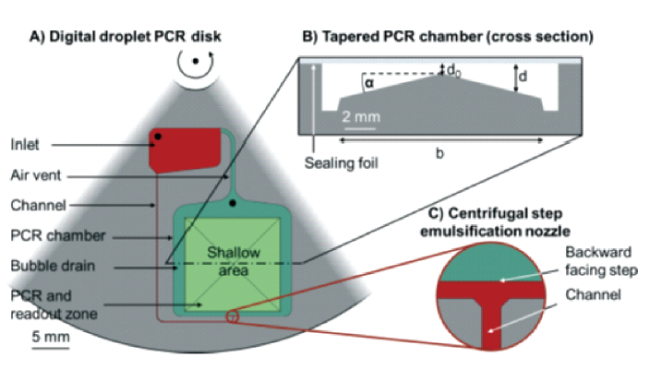
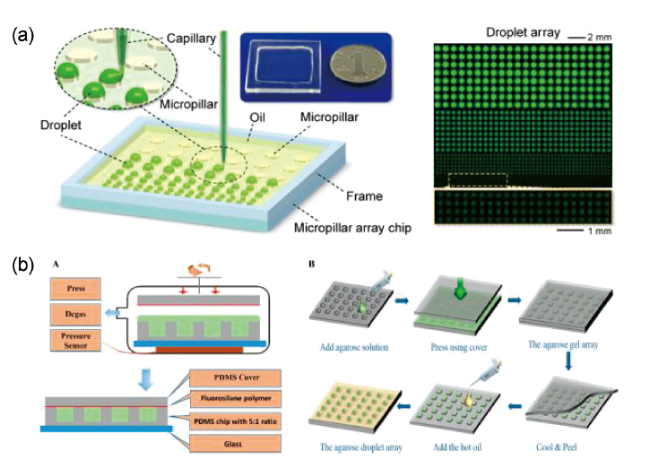
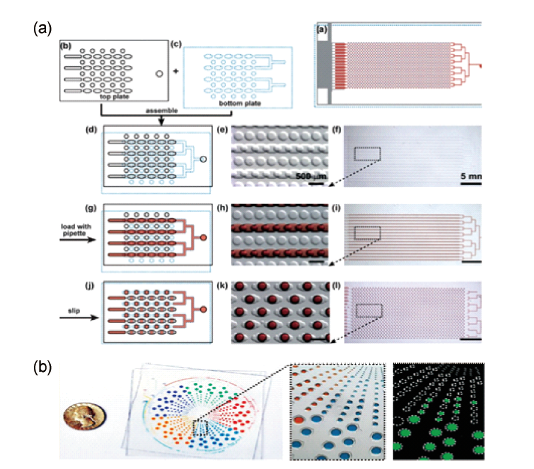
3.2 基于集成化微流控芯片的数字PCR系统
图5 (a)具有百万个皮升级微反应室的数字PCR芯片[21];(b)自分散数字PCR芯片[23];(c)自吸分液式数字PCR芯片[24];(d)PET旋转圆盘数字PCR芯片[25]Fig. 5 (a)Digital PCR chip with millions of picoliters microreaction chambers[21];(b)self-digitization digital PCR chip[23];(c)self-priming compartmentalization digital PCR chip[24];(d)PET spinning disk digital PCR chip[25] |
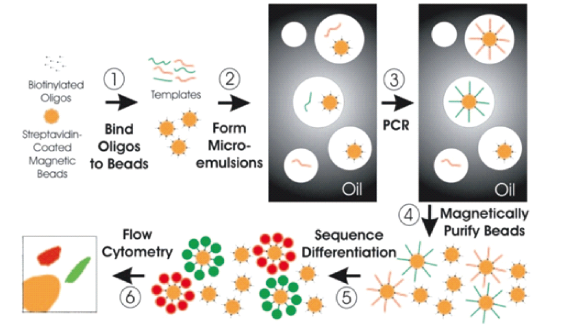
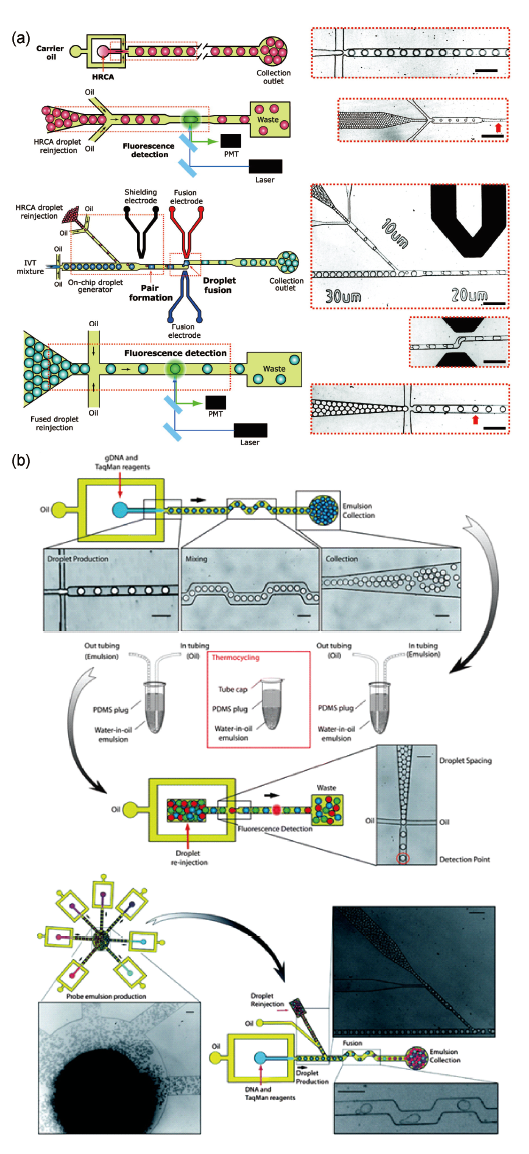
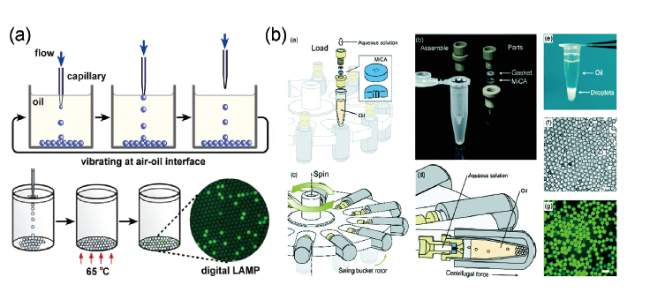
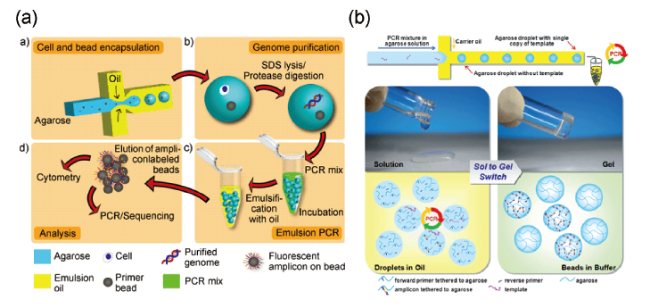
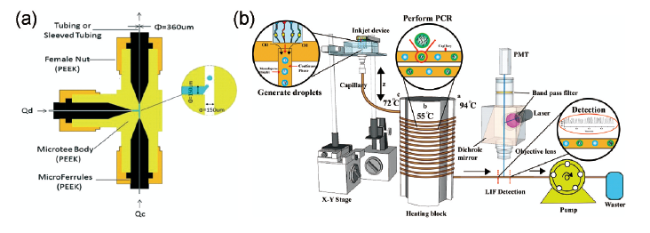
3.3 基于液滴的数字PCR系统
表1 部分商品化数字PCR仪器性能对比[51]Table 1 Performance comparison of some commercial digital PCR instruments[51] |
| Classification | Instrument | Sample dispersion | Sample consumption | Single sample reaction unit | Operating throughput | Reaction unit volume | Reaction time | Detection channel |
|---|---|---|---|---|---|---|---|---|
| Micro- chamber | ClarityTM digital PCR system | Microplate isolation | 15 μL | 10 000 | 96 | 1.5 nL | Sample loading 1 min PCR ~ 2 h | 2 |
| Quant studioTM 3D digital PCR system | Microplate isolation | 14.5 μL | 20 000 | 24 | 0.86 nL | Sample loading 4 min PCR ~2.5 h | 2 | |
| Constellation digital PCR system | Microplate isolation | 10 μL | 496 | 96 | 4 nL | Sample loading 12 min PCR ~1 h | 4 | |
| Microfluidic Chip | BioMarkTM HD High-throughput genetic analysis system | Microvalve | 8 μL | 765 | 12 | 6 nL | Sample loading 10 min PCR ~2 h | ~5 |
| 4 μL | 770 | 48 | 0.85 nL | Sample loading 40 min PCR ~2 h | ||||
| Droplet | QX200TMMicrodroplet digital PCR system | Flow-focusing | 18 μL | 20000 | 96 | 0.85 nL | Droplet generation 2 min PCR ~2 h | 2 |
| RaindropTM digital PCR system | Flow-focusing | 25~50 μL | Up to 1 000 000 | 8 | 5 pL | Droplet generation 20~30 min PCR ~3 h | 2 | |
| NaicaTM droplet digital PCR system | Step emulsification | 20 μL | 25 000~ 30 000 | 4 | 0.43 nL | Droplet generation 15 min PCR ~1 h | 3 |