1 引言
2 微流控芯片中进行血管体外重构的方法
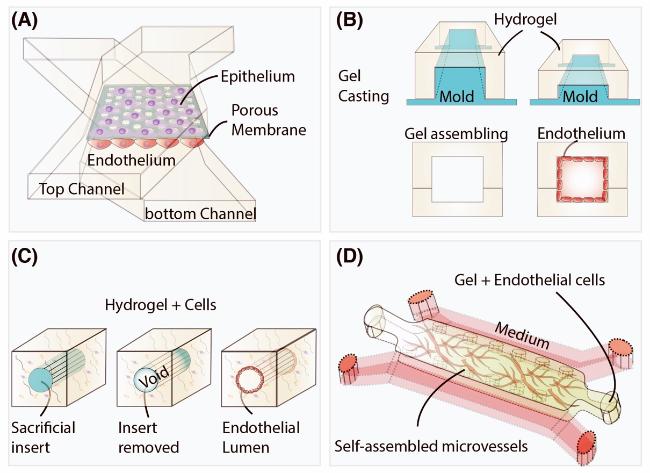
图1 体外构建血管以及血管网络用于在体外模拟类似于体内的血管和其他组织的方法归纳:(A) 利用多孔膜和与之配合的多层微流控管道构建二维单层培养的血管内皮细胞层,通常可以用来模拟血管屏障;(B) 利用微加工的模具(如聚二甲基硅氧烷(PDMS)等)将水凝胶类高分子通过塑形成为带有管腔的水凝胶微流控管道,并将血管内皮细胞通过灌注贴壁的方式培养在其中;(C) 利用打印或预先布置可牺牲凝胶或者模具的方式在凝胶中形成中空管道,并利用内皮细胞贴壁生长在凝胶中形成血管;(D) 通过将血管内皮细胞和其他支持细胞直接混合于水凝胶中,直接生长形成了毛细血管微网络。在芯片中,通过限制性结构分隔凝胶和培养基,实现对微环境的精确控制Fig. 1 Schematic illustration of in vitro construction of blood vessels and vascular networks to simulate in vivo-like vessels. (A) The vascular endothelial layer is constructed using a porous membrane in conjunction with multilayer microfluidic channels, typically used to simulate the vascular barrier. (B) Using soft lithographically fabricated molds (such as PDMS), hydrogel polymers are cast to form hydrogel microfluidic channels with lumens, and vascular endothelial cells are cultured within them by perfusion. (C) Hollow channels are formed in the gel using printing or pre-arranged sacrificial gels or molds. After the removal of the mold or gel, the endothelial cells are cultured on the walls of these channels within the gel to form vessels. (D) Vascular endothelial cells and other supporting cells are directly mixed into the hydrogel and allowed to grow within the gel to form a capillary micro-network. Functional structures like micro-pillars are used to separate the gel and culture medium in different channels of the chip |
2.1 微流控芯片中二维平面上直接培养血管内皮细胞形成单层屏障
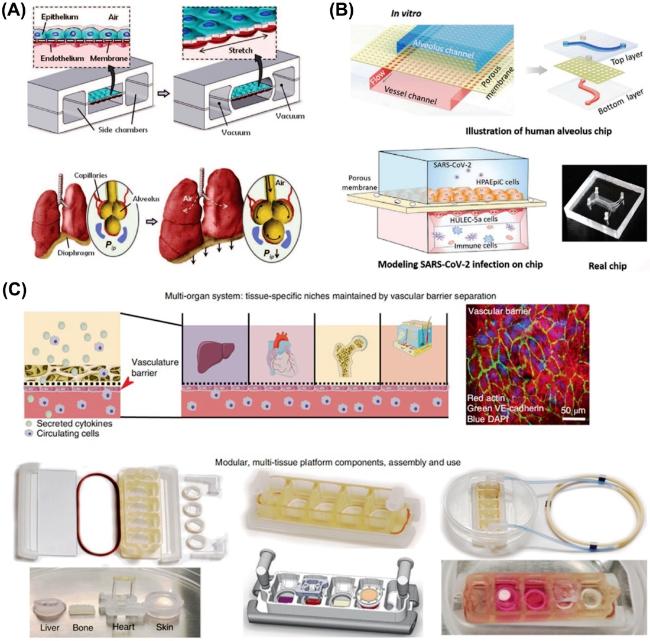
图2 基于多孔膜构建二维内皮屏障功能的微流控芯片模型:(A) 集成了弹性多孔膜以及侧翼抽气管道的微流控芯片构建和模拟肺泡-毛细血管屏障的生理呼吸过程[27];(B)受SARS-CoV-2感染的人肺泡芯片模型用于研究SARS-CoV-2病毒对人肺损伤过程和器官水平上的免疫反应[29];(C)利用多孔膜形成的血管屏障应用于多器官串联微生理系统,允许器官间的通讯与相互作用[30]Fig. 2 Recapitulation of 2D endothelial barrier functions by using the porous membrane with the microfluidic device. (A) Biological-inspired design of a human breathing lung-on-a-chip. A microfluidic chip integrates compartmentalized PDMS microchannels and an ECM-coated elastic porous membrane to form an alveolar-capillary barrier[27]. (B) A human alveolar chip model infected with SARS-CoV-2 is used to study the process of lung damage caused by the SARS-CoV-2 virus and the immune response at the organ level[29]. (C) The vascular barrier formed by the porous membrane is applied to a multi-organ linked micro-physiological system, allowing communication and interaction between organs[30] |
2.2 利用凝胶微塑形的中空管道中贴壁培养内皮细胞形成血管
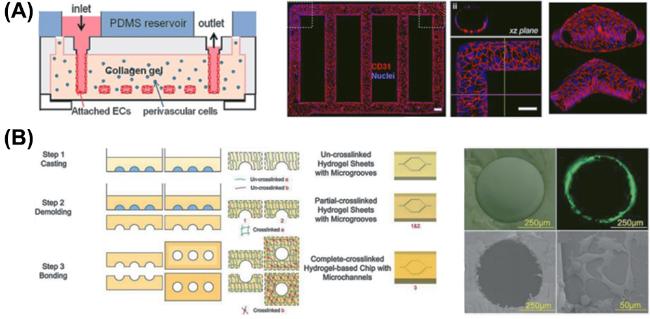
图3 利用凝胶微塑形的中空管道贴壁培养内皮细胞形成血管:(A)在胶原中微通道中接种内皮细胞形成具有完整管腔结构的血管网络并用于研究血管生成(angiogenesis)和血栓形成[32];(B)基于一种水凝胶的双交联机质在体外形成血管腔的制备工艺原理及其机理[33]Fig. 3 Using gel micro-molding to create hollow channels and culture of endothelial cells to form blood vessels. (A) Endothelial cells are seeded in microchannels within collagen to form a vascular network with a complete lumen structure, which is used to study angiogenesis and thrombosis formation[32]. (B) The principle and mechanism of the preparation process for forming vascular lumens in vitro based on a hydrogel with a dual crosslinking mechanism[33] |
2.3 利用可牺牲模具或凝胶形成管腔后贴壁培养内皮细胞形成血管
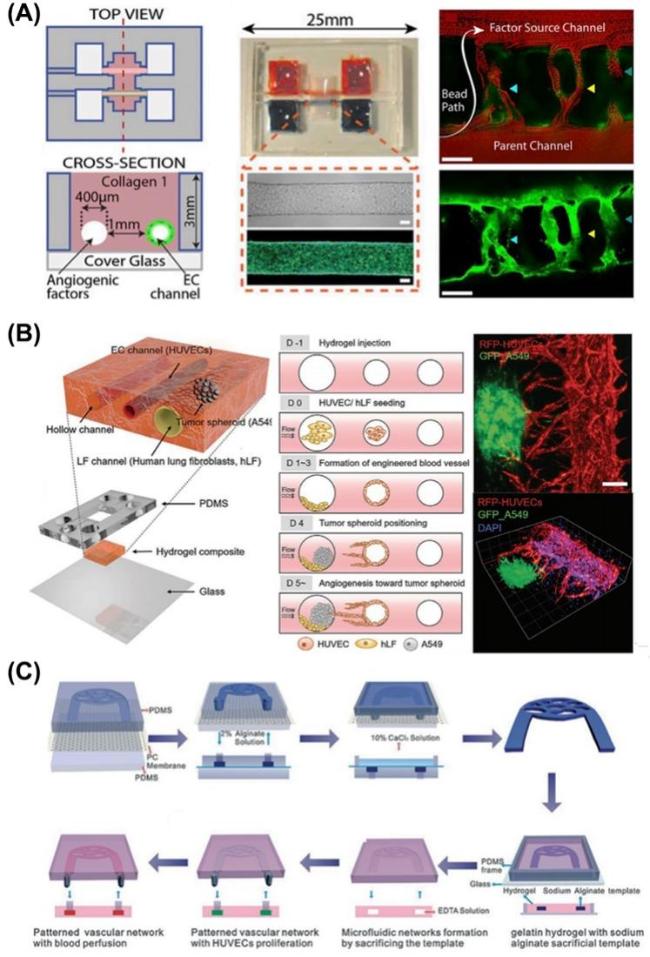
图4 利用可牺牲模具或凝胶形成管腔后贴壁培养内皮细胞形成血管:(A)基于微针去除法在体外重建血管模型用于研究新生血管芽形态发生以及相关的影响因素[37];(B)基于微针去除法在体外形成血管化肺癌肿瘤模型用于研究肿瘤微环境对血管生成的影响并评估新血管生成对促进抗癌药物给药效果[39];(C)海藻酸钠作为可牺牲模板在凝胶中快速形成相互连接的三维血管网络[41]Fig. 4 Formation of blood vessels by culturing of endothelial cells after the creation of lumens using sacrificial molds or gels inside ECM hydrogel. (A) A vascular model is reconstructed in vitro based on the microneedle removal method for studying the morphogenesis of neovascular sprouts and the related influencing factors[37]. (B) A vascularized lung cancer tumor model is formed in vitro using the microneedle removal method to study the impact of the tumor microenvironment on angiogenesis and to evaluate the effectiveness of neovascularization in enhancing the delivery of anti-cancer drugs[39]. (C) Sodium alginate is used as a sacrificial template to rapidly form interconnected three-dimensional vascular networks within a gelatin hydrogel[41] |
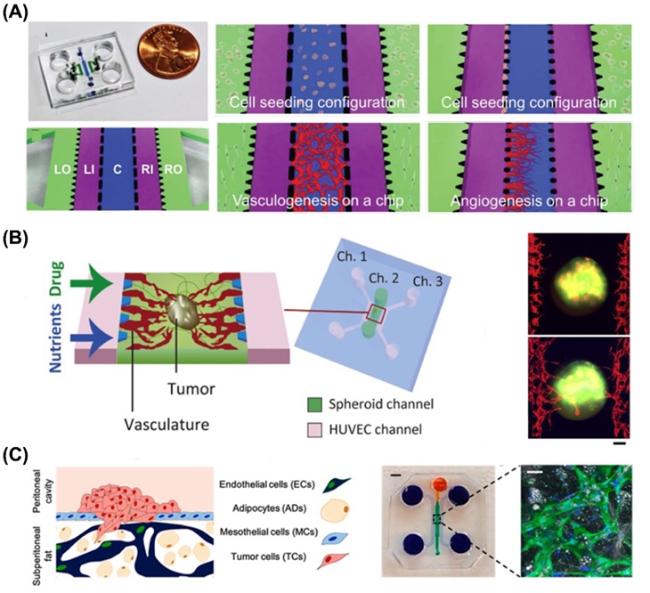
2.4 微流控芯片胶原中直接培养细胞自发形成微血管网络
图5 基于内皮细胞与基质细胞自组织的方式在凝胶中自发形成血管网络:(A)基于内皮细胞与间质成纤维细胞在微流控可控的并列管道中共培养形成完整的三维微血管,模拟正常的血管生成过程[46];(B)体外血管化的人腹膜大网膜和卵巢肿瘤微环境微流控三维模型,用于研究卵巢癌转移中基质细胞对肿瘤细胞附着和生长的影响,以及基质细胞对早期和晚期转移瘤模型中血管和间皮通透性的影响[55];(C)基于微流控芯片在体外形成血管化肿瘤模型用于研究血管对肿瘤球状体生长和药物传递的影响[56]Fig. 5 Spontaneous self-assembly formation of vascular networks within a gel based on the self-organization of endothelial cells and stromal cells. (A) The formation of complete three-dimensional microvessels through the co-culture of endothelial cells and fibroblasts within microfluidic parallel channels simulates the normal process of angiogenesis [46]. (B) The formation of a vascularized tumor model in vitro based on a microfluidic chip for studying the impact of vasculature on the growth of tumor spheroids and drug delivery [55]. (C) An in vitro vascularized human omentum and ovarian tumor microenvironment microfluidic 3D model for studying the effects of stromal cells on the attachment and growth of tumor cells during ovarian cancer metastasis, as well as the impact of stromal cells on vascular and mesothelial permeability in early and late-stage metastatic tumor models [56] |