

蛋白质凝聚作用在神经退行性疾病中的作用机制研究
收稿日期: 2022-05-04
修回日期: 2022-06-30
网络出版日期: 2022-07-15
基金资助
国家自然科学基金项目(32071212)
国家自然科学基金项目(31770833)
国家自然科学基金项目(31570779)
中国博士后科学基金项目(2021TQ0252)
中国博士后科学基金项目(2021M700103)
The Mechanism of Protein Condensation in Neurodegenerative Diseases
Received date: 2022-05-04
Revised date: 2022-06-30
Online published: 2022-07-15
Supported by
National Natural Science Foundation of China(32071212)
National Natural Science Foundation of China(31770833)
National Natural Science Foundation of China(31570779)
China Postdoctoral Science Foundation(2021TQ0252)
China Postdoctoral Science Foundation(2021M700103)
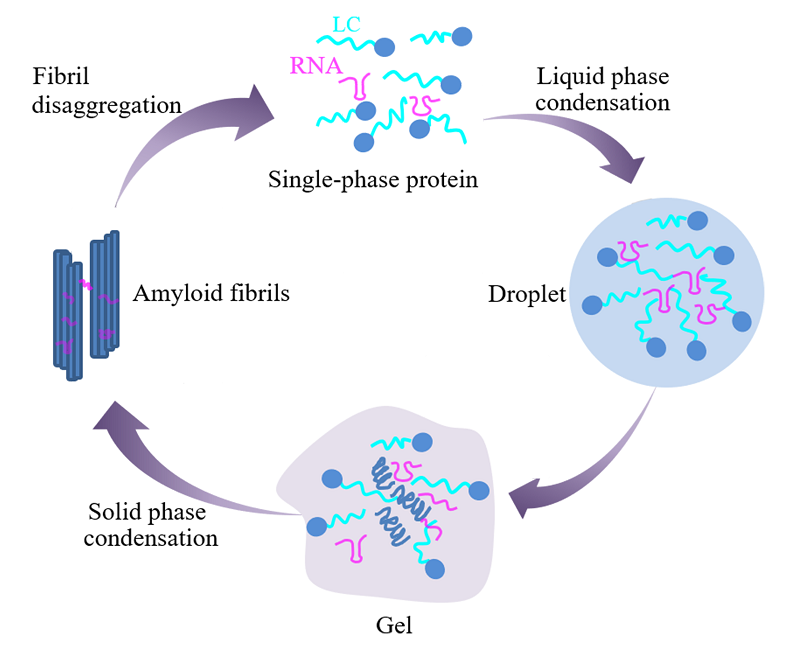
蛋白质和RNA通过液-液相分离组装成无膜细胞器。无膜细胞器与液滴具有相似的融合性质,当浓度超过饱和浓度时,生物大分子会形成液滴,接着向凝胶态进行转化,最终形成固态凝聚体。传染性海绵状脑病、肌萎缩侧索硬化症和阿尔茨海默病等神经退行性疾病共同的病理特征是,错误折叠的蛋白质(包括朊蛋白、TDP-43和Tau蛋白)形成有毒性的寡聚体或淀粉样纤维。大量研究表明,这些蛋白质都可以发生液-液相分离形成凝聚体。本文综述了蛋白质凝聚作用在传染性海绵状脑病、TDP-43蛋白病以及 Tau蛋白病中的作用机制,重点阐述了相分离如何诱导神经退行性疾病中错误折叠朊蛋白、TDP-43和Tau蛋白形成寡聚体和淀粉样纤维,并讨论和展望了蛋白质凝聚作用与神经退行性疾病关联研究中存在的挑战和机遇。
张沐雅 , 刘嘉琪 , 陈旺 , 王利强 , 陈杰 , 梁毅 . 蛋白质凝聚作用在神经退行性疾病中的作用机制研究[J]. 化学进展, 2022 , 34(7) : 1619 -1625 . DOI: 10.7536/PC220534
Protein and RNA molecules tend to form supramolecular assemblies called membrane-less organelles via liquid-liquid phase separation of proteins in cells. These organelles have fusion properties similar to liquid droplets formed by biological macromolecules when their concentrations are higher than saturation concentrations. Upon aging, these dynamic droplets change their material properties and transform into gels, followed by formation of solid condensates. It is well known that proteins with low-complexity domains undergo liquid-liquid phase separation. The common pathological feature of neurodegenerative diseases such as transmissible spongiform encephalopathy, amyotrophic lateral sclerosis, and Alzheimer’s disease is toxic oligomers or amyloid aggregates formed by misfolded proteins including prion protein, DNA- and RNA-binding protein TDP-43, and Tau protein. A large number of studies have shown that prion protein, TDP-43, and Tau protein all undergo liquid-liquid phase separation and form protein condensates. This review summarizes the role of protein phase separation and condensation in neurodegenerative diseases, elaborates the mechanism of protein condensation modulating transmissible spongiform encephalopathies, TDP-43 proteinopathies, and tauopathies, and focuses on the initiation effect of phase separation on aggregation and toxicity of misfolded proteins in neurodegenerative diseases. Finally, we discuss and prospect the challenges and opportunities of association study between protein condensation and neurodegenerative diseases.
1 Introduction
2 Liquid-liquid phase separation and condensation of prion proteins
3 Liquid-liquid phase separation and condensation of TDP-43
4 Liquid-liquid phase separation and condensation of Tau protein
5 Discussion
6 Challenge and outlook

Key words: protein phase separation; neurodegenerative diseases; prion protein; TDP-43; Tau; condensate
| [1] |
Palomo G M, Manfredi G. Brain Res., 2015, 1607: 36.
|
| [2] |
Weber S C, Brangwynne C P. Cell, 2012, 149(6): 1188.
|
| [3] |
Mitrea D M, Kriwacki R W. Cell Commun. Signal., 2016, 14: 1.
|
| [4] |
Bergeron-Sandoval L P, Safaee N, Michnick S W. Cell, 2016, 165(5): 1067.
|
| [5] |
Hong K, Song D, Jung Y. Nat. Commun., 2020, 11: 5554.
|
| [6] |
Boeynaems S, Alberti S, Fawzi N L, Mittag T, Polymenidou M, Rousseau F, Schymkowitz J, Shorter J, Wolozin B, van den Bosch L, Tompa P, Fuxreiter M. Trends Cell Biol., 2018, 28(6): 420.
|
| [7] |
Brangwynne C P, Eckmann C R, Courson D S, Rybarska A, Hoege C, Gharakhani J, Jülicher F, Hyman A A. Science, 2009, 324(5935): 1729.
|
| [8] |
Shimobayashi S F, Ronceray P, Sanders D W, Haataja M P, Brangwynne C P. Nature, 2021, 599(7885): 503.
|
| [9] |
Shin Y, Brangwynne C P. Science, 2017, 357(6357): eaaf4382.
|
| [10] |
Molliex A, Temirov J, Lee J H, Coughlin M, Kanagaraj A P, Kim H J, Mittag T, Taylor J P. Cell, 2015, 163(1): 123.
|
| [11] |
Wegmann S, Eftekharzadeh B, Tepper K, Zoltowska K M, Bennett R E, Dujardin S, Laskowski P R, MacKenzie D, Kamath T, Commins C, Vanderburg C, Roe A D, Fan Z Y, Molliex A M, Hernandez-Vega A, Muller D, Hyman A A, Mandelkow E, Taylor J P, Hyman B T. EMBO J., 2018, 37(7): e98049.
|
| [12] |
Mathieu C, Pappu R V, Taylor J P. Science, 2020, 370(6512): 56.
|
| [13] |
Banani S F, Lee H O, Hyman A A, Rosen M K. Nat. Rev. Mol. Cell Biol., 2017, 18(5): 285.
|
| [14] |
Scheckel C, Aguzzi A. Nat. Rev. Genet., 2018, 19(7): 405.
|
| [15] |
Ayers J I, Borchelt D R. Acta Neuropathol., 2021, 142(1): 41.
|
| [16] |
Wang Y P, Mandelkow E. Nat. Rev. Neurosci., 2016, 17(1): 22.
|
| [17] |
Agarwal A, Mukhopadhyay S. J. Mol. Biol., 2022, 434(1): 167368.
|
| [18] |
Kostylev M A, Tuttle M D, Lee S, Klein L E, Takahashi H, Cox T O, Gunther E C, Zilm K W, Strittmatter S M. Mol. Cell, 2018, 72(3): 426.
|
| [19] |
Huang J J, Li X N, Liu W L, Yuan H Y, Gao Y, Wang K, Tang B, Pang D W, Chen J, Liang Y. J. Mol. Biol., 2020, 432(4): 828.
|
| [20] |
Matos C O, Passos Y M, Amaral M J, Macedo B, Tempone M H, Bezerra O C L, Moraes M O, Almeida M S, Weber G, Missailidis S, Silva J L, Uversky V N, Pinheiro A S, Cordeiro Y. FASEB J., 2020, 34(1): 365.
|
| [21] |
Tange H, Ishibashi D, Nakagaki T, Taguchi Y, Kamatari Y O, Ozawa H, Nishida N. J. Biol. Chem., 2021, 296: 100367.
|
| [22] |
Agarwal A, Rai S K, Avni A, Mukhopadhyay S. PNAS, 2021, 118(45): e2100968118.
|
| [23] |
M Passos Y, J do Amaral M, C Ferreira N, Macedo B, Chaves J A P, de Oliveira V E, P B Gomes M, L Silva J, Cordeiro Y. Int. J. Biol. Macromol., 2021, 173: 34.
|
| [24] |
Kamps J, Lin Y H, Oliva R, Bader V, Winter R, Winklhofer K F, Tatzelt J. J. Biol. Chem., 2021, 297(1): 100860.
|
| [25] |
Agarwal A, Arora L, Rai S K, Avni A, Mukhopadhyay S. Nat. Commun., 2022, 13: 1154.
|
| [26] |
Li H R, Chiang W C, Chou P C, Wang W J, Huang J R. J. Biol. Chem., 2018, 293(16): 6090.
|
| [27] |
Hallegger M, Chakrabarti A M, Lee F C Y, Lee B L, Amalietti A G, Odeh H M, Copley K E, Rubien J D, Portz B, Kuret K, Huppertz I, Rau F, Patani R, Fawzi N L, Shorter J, Luscombe N M, Ule J. Cell, 2021, 184(18): 4680.
|
| [28] |
Conicella A E, Dignon G L, Zerze G H, Schmidt H B, D’Ordine A M, Kim Y C, Rohatgi R, Ayala Y M, Mittal J, Fawzi N L. PNAS, 2020, 117(11): 5883.
|
| [29] |
Pantoja-Uceda D, Stuani C, Laurents D V, McDermott A E, Buratti E, Mompeán M. PLoS Biol., 2021, 19(4): e3001198.
|
| [30] |
Wang A L, Conicella A E, Schmidt H B, Martin E W, Rhoads S N, Reeb A N, Nourse A, Ramirez Montero D, Ryan V H, Rohatgi R, Shewmaker F, Naik M T, Mittag T, Ayala Y M, Fawzi N L. EMBO J., 2018, 37(5): e97452.
|
| [31] |
Wang C, Duan Y, Duan G, Wang Q, Zhang K, Deng X, Qian B, Gu J, Ma Z, Zhang S, Guo L, Liu C, Fang Y. Mol. Cell, 2020, 79(3): 443.
|
| [32] |
Gasset-Rosa F, Lu S, Yu H Y, Chen C, Melamed Z, Guo L, Shorter J, da Cruz S, Cleveland D W. Neuron, 2019, 102(2): 339.
|
| [33] |
Yu H, Lu S, Gasior K, Singh D, Vazquez-Sanchez S, Tapia O, Toprani D, Beccari M S, Yates J R 3rd, Da Cruz S, Newby J M, Lafarga M, Gladfelter A S, Villa E, Cleveland D W. Science, 2021, 371(6529): eabb4309.
|
| [34] |
MacKenzie I R, Nicholson A M, Sarkar M, Messing J, Purice M D, Pottier C, Annu K, Baker M, Perkerson R B, Kurti A, Matchett B J, Mittag T, Temirov J, Hsiung G Y R, Krieger C, Murray M E, Kato M, Fryer J D, Petrucelli L, Zinman L, Weintraub S, Mesulam M, Keith J, Zivkovic S A, Hirsch-Reinshagen V, Roos R P, Züchner S, Graff-Radford N R, Petersen R C, Caselli R J, Wszolek Z K, Finger E, Lippa C, Lacomis D, Stewart H, Dickson D W, Kim H J, Rogaeva E, Bigio E, Boylan K B, Taylor J P, Rademakers R. Neuron, 2017, 95(4): 808.
|
| [35] |
Krainer G, Welsh T J, Joseph J A, Espinosa J R, Wittmann S, Sridhar A, Toprakcioglu Z, Gudiškyt?G, Czekalska M A, Arter W E, GuillÉn-Boixet J, Franzmann T M, Qamar S, George-Hyslop P S, Hyman A A, Collepardo-Guevara R, Alberti S, Knowles T P J. Nat. Commun., 2021, 12: 1085.
|
| [36] |
da Silva L A G, Simonetti F, Hutten S, Riemenschneider H, Sternburg E L, Pietrek L M, Gebel J, Dötsch V, Edbauer D, Hummer G, Stelzl L S, Dormann D. EMBO J., 2022, 41(8): e108443.
|
| [37] |
Garcia Morato J, Hans F, von Zweydorf F, Feederle R, Elsässer S J, Skodras A A, Gloeckner C J, Buratti E, Neumann M, Kahle P J. Nat. Commun., 2022, 13: 1223.
|
| [38] |
Duan Y J, Du A Y, Gu J G, Duan G, Wang C, Gui X R, Ma Z W, Qian B T, Deng X, Zhang K, Sun L, Tian K L, Zhang Y Y, Jiang H, Liu C, Fang Y S. Cell Res., 2019, 29(3): 233.
|
| [39] |
McGurk L, Gomes E, Guo L, Mojsilovic-Petrovic J, Tran V, Kalb R G, Shorter J, Bonini N M. Mol. Cell, 2018, 71(5): 703.
|
| [40] |
Fang M Y, Markmiller S, Vu A Q, Javaherian A, Dowdle W E, Jolivet P, Bushway P J, Castello N A, Baral A, Chan M Y, Linsley J W, Linsley D, Mercola M, Finkbeiner S, Lecuyer E, Lewcock J W, Yeo G W. Neuron, 2019, 103(5): 802.
|
| [41] |
Babinchak W M, Dumm B K, Venus S, Boyko S, Putnam A A, Jankowsky E, Surewicz W K. Nat. Commun., 2020, 11: 5574.
|
| [42] |
Boyko S, Surewicz K, Surewicz W K. PNAS, 2020, 117(50): 31882.
|
| [43] |
Ambadipudi S, Biernat J, Riedel D, Mandelkow E, Zweckstetter M. Nat. Commun., 2017, 8: 275.
|
| [44] |
Ambadipudi S, Reddy J G, Biernat J, Mandelkow E, Zweckstetter M. Chem. Sci., 2019, 10(26): 6503.
|
| [45] |
Lin Y X, Fichou Y, Zeng Z K, Hu N Y, Han S. ACS Chem. Neurosci., 2020, 11(4): 615.
|
| [46] |
Ferreon J, Jain A, Choi K J, Tsoi P, MacKenzie K, Jung S, Ferreon A. Int. J. Mol. Sci., 2018, 19(5): 1360.
|
| [47] |
Lin Y, McCarty J, Rauch J N, Delaney K T, Kosik K S, Fredrickson G H, Shea J E, Han S. Elife, 2019, 8: e42571.
|
| [48] |
Ukmar-Godec T, Hutten S, Grieshop M P, Rezaei-Ghaleh N, Cima-Omori M S, Biernat J, Mandelkow E, Söding J, Dormann D, Zweckstetter M. Nat. Commun., 2019, 10: 2909.
|
| [49] |
Rane J S, Kumari A, Panda D. ACS Chem. Neurosci., 2020, 11(3): 291.
|
| [50] |
Singh V, Xu L, Boyko S, Surewicz K, Surewicz W K. J. Biol. Chem., 2020, 295(18): 5850.
|
| [51] |
Gao Y Y, Zhong T, Wang L Q, Zhang N, Zeng Y, Hu J Y, Dang H B, Chen J, Liang Y. Int. J. Biol. Macromol., 2022, 209: 703.
|
| [52] |
Wang K, Liu J Q, Zhong T, Liu X L, Zeng Y, Qiao X H, Xie T, Chen Y Z, Gao Y Y, Tang B, Li J, Zhou J, Pang D W, Chen J, Chen C, Liang Y. J. Mol. Biol., 2020, 432(7): 2141.
|
| [53] |
Dai B, Zhong T, Chen Z X, Chen W, Zhang N, Liu X L, Wang L Q, Chen J, Liang Y. J. Biol. Chem., 2021, 297(4): 101222.
|
| [54] |
Zhang X M, Lin Y X, Eschmann N A, Zhou H J, Rauch J N, Hernandez I, Guzman E, Kosik K S, Han S. PLoS Biol., 2017, 15(7): e2002183.
|
| [55] |
Majumdar A, Dogra P, Maity S, Mukhopadhyay S. J. Phys. Chem. Lett., 2019, 10(14): 3929.
|
| [56] |
Kanaan N M, Hamel C, Grabinski T, Combs B. Nat. Commun., 2020, 11: 2809.
|
| [57] |
Jonchhe S, Pan W, Pokhrel P, Mao H B. Angewandte Chemie Int. Ed., 2022, 61(23): e202113156.
|
| [58] |
Prasad A, Bharathi V, Sivalingam V, Girdhar A, Patel B K. Front. Mol. Neurosci., 2019, 12: 25.
|
| [59] |
Verdile V, de Paola E, Paronetto M P. Front. Genet., 2019, 10: 173.
|
| [60] |
Prusiner S B. PNAS, 1998, 95(23): 13363.
|
| [61] |
Prusiner S B. Trends Biochem. Sci., 1996, 21(12): 482.
|
| [62] |
Pan K M, Baldwin M, Nguyen J, Gasset M, Serban A, Groth D, Mehlhorn I, Huang Z, Fletterick R J, Cohen F E. PNAS, 1993, 90(23): 10962.
|
| [63] |
Diaz-Espinoza R, Soto C. Nat. Struct. Mol. Biol., 2012, 19(4): 370.
|
| [64] |
Kraus A, Hoyt F, Schwartz C L, Hansen B, Artikis E, Hughson A G, Raymond G J, Race B, Baron G S, Caughey B. Mol. Cell, 2021, 81(21): 4540.
|
| [65] |
Wang L Q, Zhao K, Yuan H Y, Wang Q, Guan Z Y, Tao J, Li X N, Sun Y P, Yi C W, Chen J, Li D, Zhang D L, Yin P, Liu C, Liang Y. Nat. Struct. Mol. Biol., 2020, 27(6): 598.
|
| [66] |
Wang L Q, Zhao K, Yuan H Y, Li X N, Dang H B, Ma Y Y, Wang Q, Wang C, Sun Y P, Chen J, Li D, Zhang D L, Yin P, Liu C, Liang Y. Sci. Adv., 2021, 7(37): eabg9676.
|
| [67] |
Polymenidou M, Cleveland D W. Cell, 2011, 147(3): 498.
|
| [68] |
Xu W C, Liang J Z, Li C, He Z X, Yuan H Y, Huang B Y, Liu X L, Tang B, Pang D W, Du H N, Yang Y, Chen J, Wang L, Zhang M, Liang Y. Cell Death Dis., 2018, 9(2): 67.
|
| [69] |
Wang L Q, Ma Y, Yuan H Y, Zhao K, Zhang M Y, Wang Q, Huang X, Xu W C, Dai B, Chen J, Li D, Zhang D, Wang Z, Zou L, Yin P, Liu C, Liang Y. Nat. Commun., 2022, 13(1): 3491.
|
| [70] |
Buratti E, Baralle F E. Trends Biochem. Sci., 2012, 37(6): 237.
|
| [71] |
Neumann M, Sampathu D M, Kwong L K, Truax A C, Micsenyi M C, Chou T T, Bruce J, Schuck T, Grossman M, Clark C M, McCluskey L F, Miller B L, Masliah E, MacKenzie I R, Feldman H, Feiden W, Kretzschmar H A, Trojanowski J Q, Lee V M Y. Science, 2006, 314(5796): 130.
|
| [72] |
Kuo P H, Chiang C H, Wang Y T, Doudeva L G, Yuan H S. Nucleic Acids Res., 2014, 42(7): 4712.
|
| [73] |
Boyko S, Qi X, Chen T H, Surewicz K, Surewicz W K. J. Biol. Chem., 2019, 294(29): 11054.
|
/
| 〈 |
|
〉 |