1 引言
2 质谱电离方法概述
2.1 电子轰击/俘获电离
2.2 电喷雾电离和基质辅助电离
2.3 表面电离
2.4 原子/离子束电离
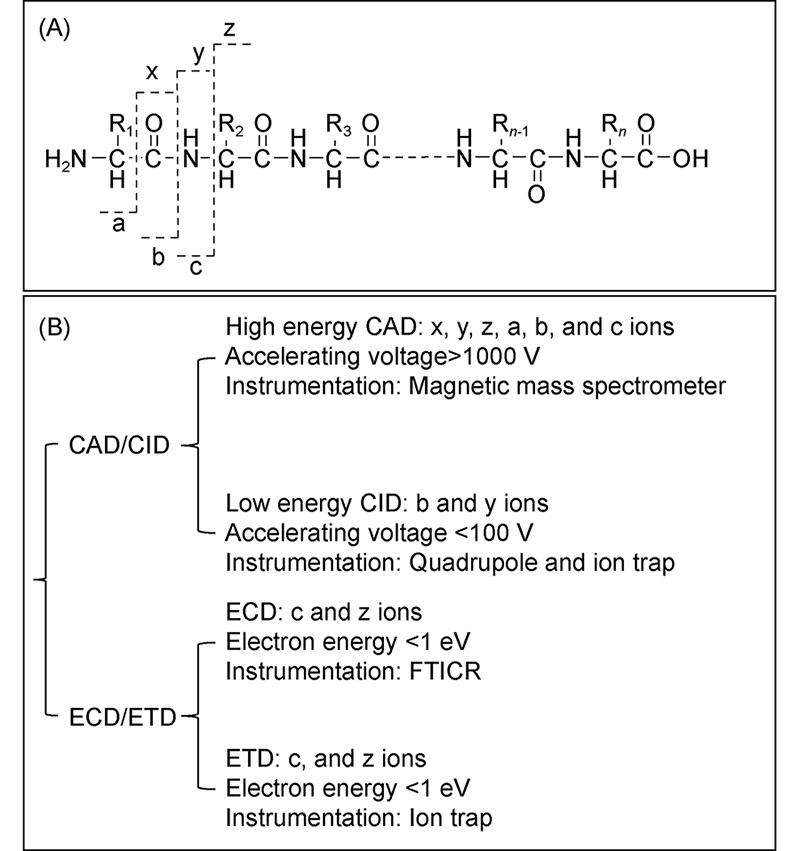
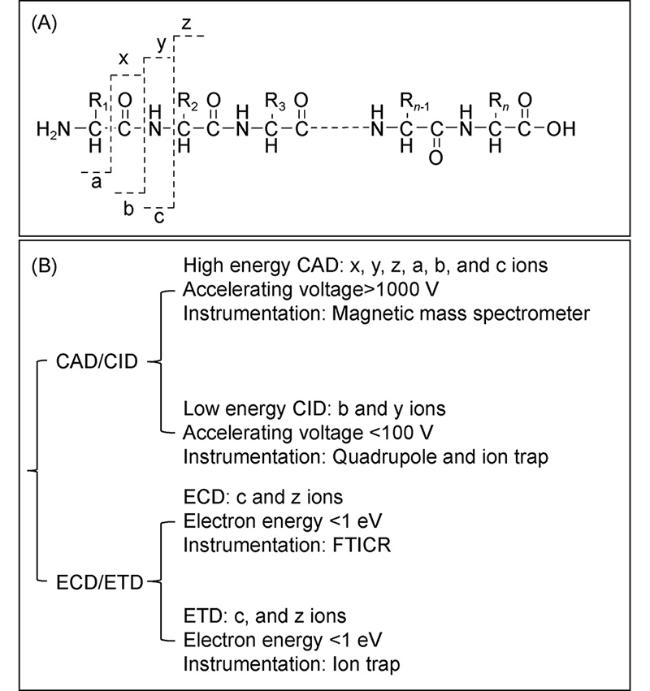
3 质谱解离技术概述
4 光电离/解离基本原理
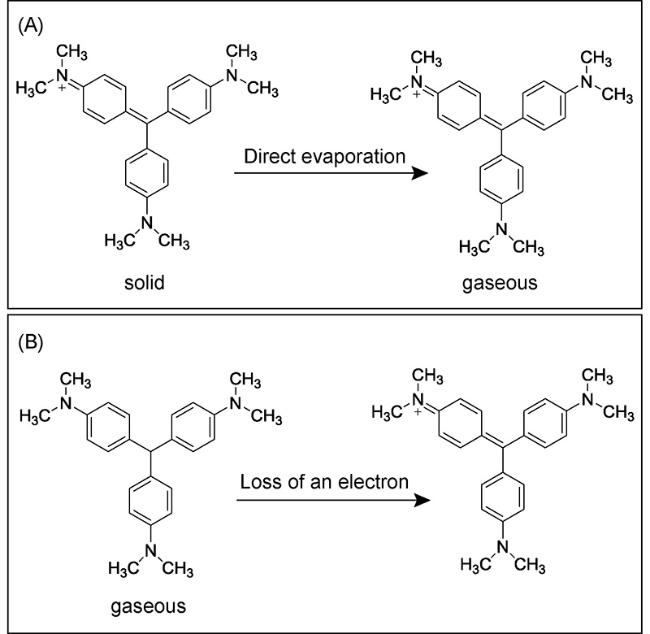
图2 激光解吸电离甲基紫的两种途径。(A)激光热效应直接气化固态甲基紫盐酸盐;(B)激光激励中性甲基紫丢失低电离电位电子Fig.2 Laser desorption dissociation of methyl violet. (A) Direct evaporation of solid methyl violet hydrochloride by laser heating effect; (B) Losses of low ionization potential electrons of neutral methyl violet by laser excitation |
4.1 直接光电离和解离
表1 质谱中不同电离技术的原理及应用Table 1 Principle and application of different ionization techniques in mass spectrometry |
| No. | Ionization | Principles and products | Molecular ions | Fragment ions | Whether the coupling to other techniques for fragmentation is needed (Y/N) | Applications | |
|---|---|---|---|---|---|---|---|
| 1 | Electron-initiated ionization | Electron impact (EI) | Loss of electrons with low ionization potential Radical cation | Ions with odd-numbered electrons | Radical/charge-initiated homolytic or heterolytic bond cleavages | N | Volatile small organic molecules |
| Laser activated electron tunneling (LAET) | Electron capture by charge deficient atoms Radical anions | Ions with odd-numbered electrons | Radical/charge-initiated homolytic or heterolytic bond cleavages | N | small organic molecules | ||
| 2 | Electrospray ionization (ESI) | Protonation/deprotonation Metal ion adducts | Ions with even-numbered electrons multiple-charged ions | Difficult to occur spontaneously | Y Collision-activated dissociation, electron transfer dissociation and photo dissociation | biological macromolecules/small organic molecules | |
| 3 | Matrix assisted laser desorption ionization (MALDI) | Protonation/deprotonation Metal ion adducts | Ions with even-numbered electrons | Difficult to occur spontaneously | Y Collision-activated dissociation and photo dissociation | biological macromolecules | |
| 4 | Surface ionization | Surface Enhanced Laser Desorption Ionization (SALDI) | Protonation/deprotonation Metal ion adducts | Ions with even-numbered electrons | Difficult to occur spontaneously | Y Collision-activated dissociation and photo dissociation | biological macromolecules/small organic molecules |
| Desorption Ionization on Porous Silicon (DIOS) | Protonation/deprotonation Metal ion adducts | Ions with even-numbered electrons | Difficult to occur spontaneously | Y Collision-activated dissociation and photo dissociation | biological macromolecules/small organic molecules | ||
| Nanostructure-Initiator Mass Spectrometry (NIMS) | Protonation/deprotonation Metal ion adducts | Ions with even-numbered electrons | Difficult to occur spontaneously | Y Collision-activated dissociation and photo dissociation | biological macromolecules/small organic molecules | ||
| 5 | Atomic/ion beam ionization | Fast Atom Bombardment (FAB) | Protonation/deprotonation Metal ion adducts | Ions with even-numbered electrons | Difficult to occur spontaneously | Y Collision-activated dissociation | Polypeptide/small organic molecules |
| Secondary Ion Mass Spectrometry (SIMS) | Positive/negative ions | Ions with even-numbered electrons | Vibration activated dissociation | N | Material elements/small organic molecules/insulators | ||
| Desorption Electrospray Ionization (DESI) | Protonation/deprotonation Metal ion adducts | Ions with even-numbered electrons multiple-charged ions | Difficult to occur spontaneously | Y Collision-activated dissociation, electron transfer dissociation and photo dissociation | biological macromolecules/small organic molecules | ||
4.2 光解离与其他电离技术的联用
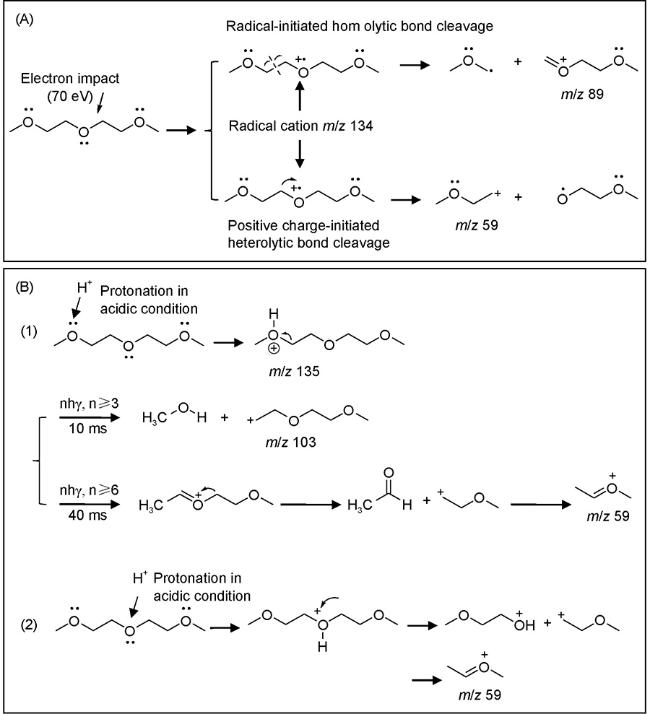
图3 红外多光子解离与电子轰击电离的比较。(A)电子轰击产生的自由基中心和正电荷中心及其引发的化学键断裂;(B)红外多光子振动激活的化学键断裂Fig.3 Comparison of infrared multiphoton dissociation and electron impact ionization. (A) Radical/charge centers generated by electron impact ionization and resultant chemical bond cleavages; (B) Infrared multiphoton activated chemical bond cleavages |
5 光解离质谱仪器的结构及特点
5.1 红外多光子解离质谱仪
5.2 紫外光解离质谱仪
6 生物分子光解离及结构鉴定
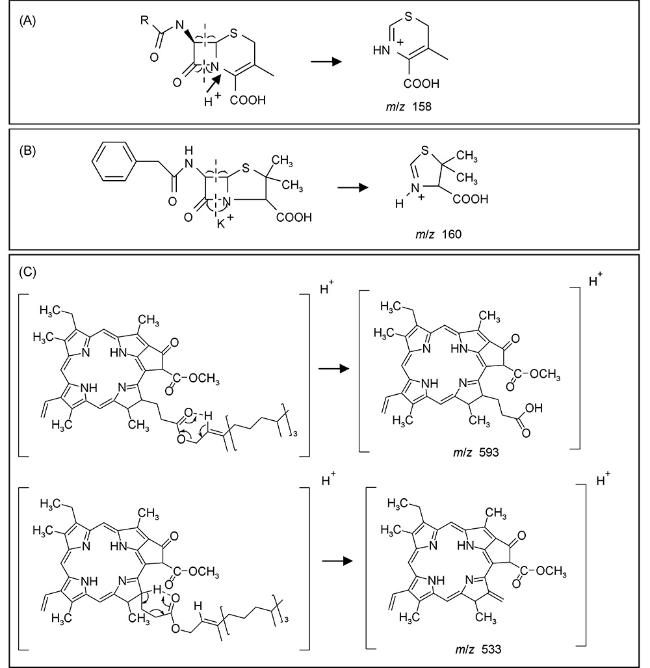
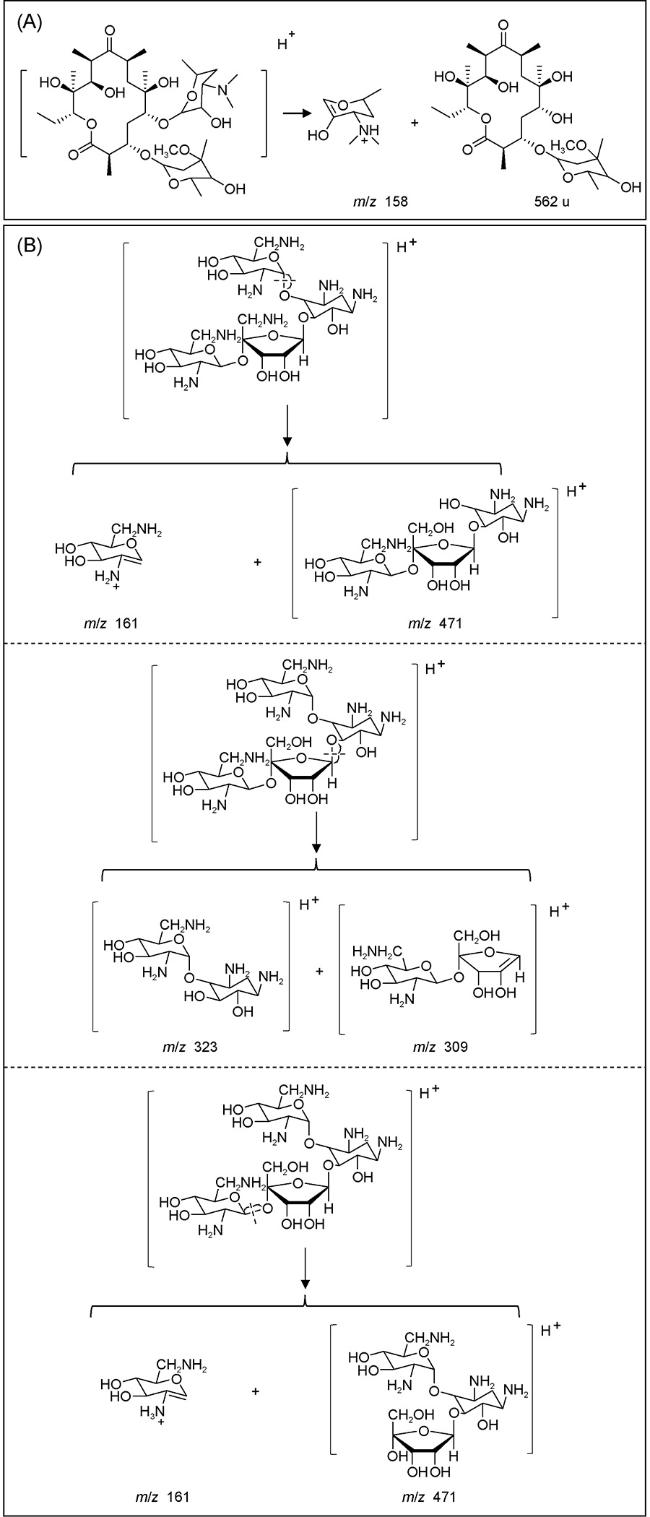
6.1 有机小分子结构鉴定
 O和C—O键均可由红外激光引发化学键振动激活。
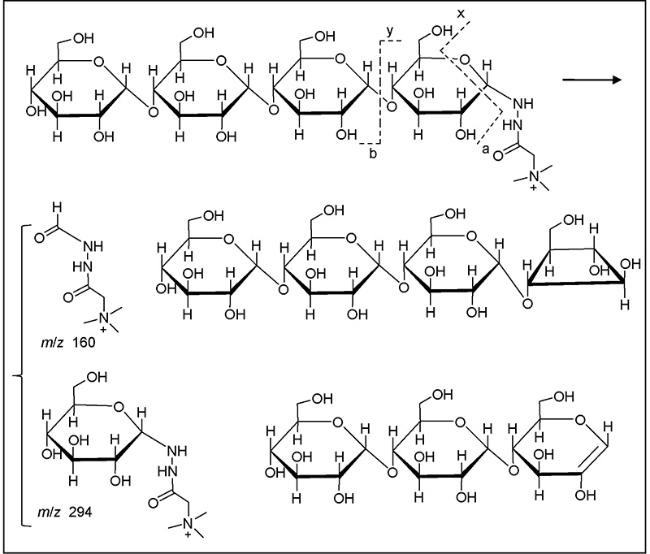
O和C—O键均可由红外激光引发化学键振动激活。6.2 单糖和多糖结构鉴定
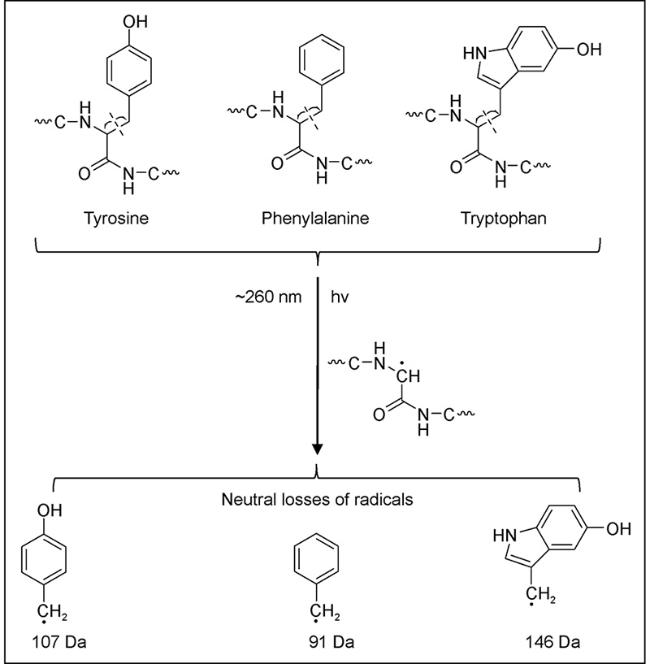
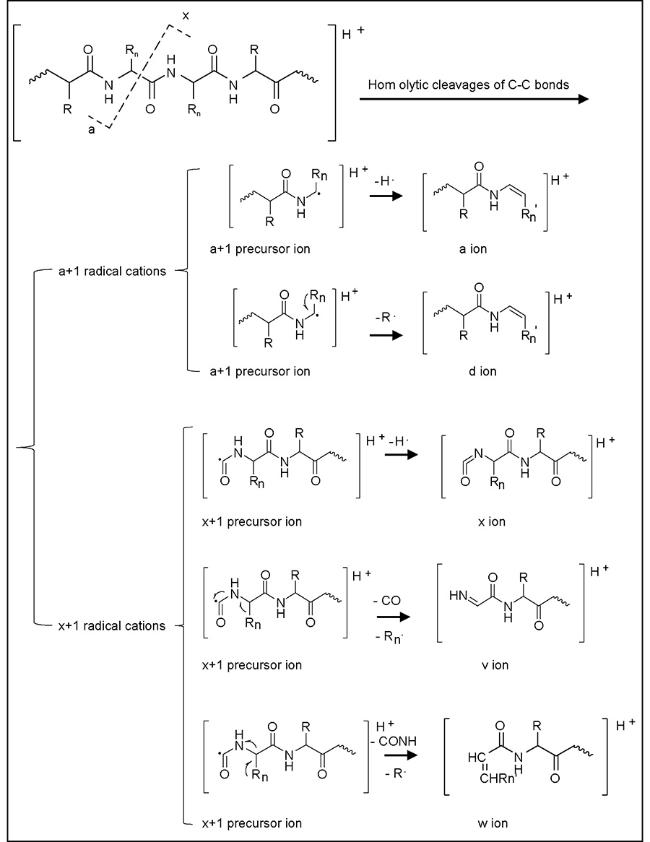
6.3 多肽/蛋白质氨基酸序列和翻译后修饰鉴定
 Cβ 键能够与酰胺键共轭,结构更加稳定。一般情况下,d 离子不会出现在无 β—C 取代的甘氨酸和丙氨酸残基中,而消除芳香环需要更高能量,所以芳香氨基酸残基也不会产生d离子[165]。当利用紫外光去解离ECD和ETD产生的气态离子时,如果精氨酸在N端,则主要产生a离子,符合精氨酸效应。但如果精氨酸处于C端,则会断裂N-Cα键形成c和z离子[169]。离子解离也与电荷定位有关,如H+和Na+定位于碱性氨基酸残基,容易发生电荷引发的化学键断裂[170]。
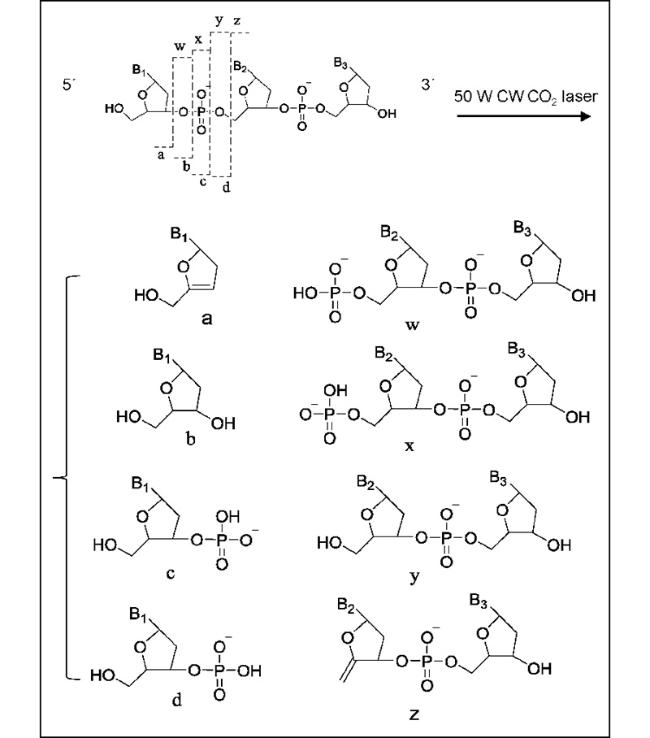
Cβ 键能够与酰胺键共轭,结构更加稳定。一般情况下,d 离子不会出现在无 β—C 取代的甘氨酸和丙氨酸残基中,而消除芳香环需要更高能量,所以芳香氨基酸残基也不会产生d离子[165]。当利用紫外光去解离ECD和ETD产生的气态离子时,如果精氨酸在N端,则主要产生a离子,符合精氨酸效应。但如果精氨酸处于C端,则会断裂N-Cα键形成c和z离子[169]。离子解离也与电荷定位有关,如H+和Na+定位于碱性氨基酸残基,容易发生电荷引发的化学键断裂[170]。6.4 核酸序列分析
7 结论与展望
表2 通过光电离和解离对生物分子的质谱鉴定Table 2 Mass spectrometric identification of biological molecules with photo ionization and dissociation |
| Samples | IR1 | UV2 | ||||
|---|---|---|---|---|---|---|
| Principles | Features | Disadvantages | Principles | Features | Disadvantages | |
| Small organic molecules | Bond cleavages by vibrational excitation | Selective absorption in IR region by different bonds | Need multiple IR photons to activate bond cleavages due to the low energy of each IR photon | Electron/charge-directed bond cleavages + vibration excited bond cleavages | Do not need multiple UV photons | Extensive bond cleavages/Need chemical derivatization in the near UV light region |
| Monosaccharides/polysaccharides | Distinguish O-, N-, C- and S- glycosidic bonds | ·Characteristic a, b, y and x ions as well as neutral losses in the vacuum UV region. | ||||
| Peptides/Proteins | ·Characteristic b and y ions resulting from C-N bond cleavages. ·Identification of fragile posttranslational modification that is difficult in CAD3. | ·Selective cleavages of S-S bonds in the near UV region. ·Characteristic a, x, d, v and w ions in the vacuum UV region. ·Arginine effects on the production of a or x ions | ||||
1Infrared (IR),2Ultraviolet (UV),3Collision activated dissociation (CAD) |