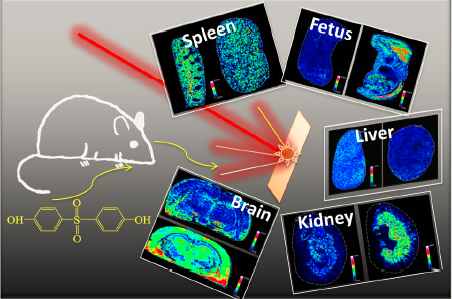
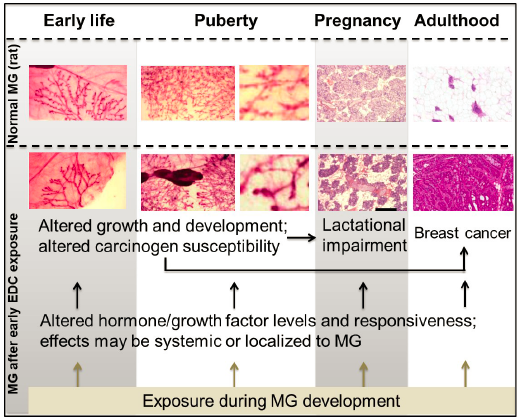
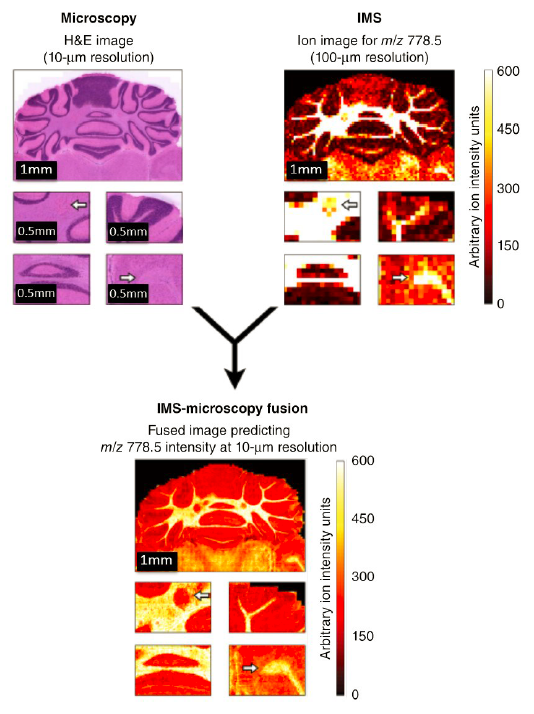
1 引言
2 MSI和组学分析
2.1 污染物暴露的模型构建
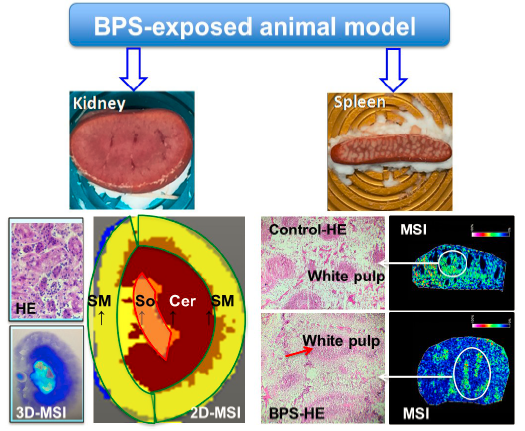
2.2 污染物暴露的组织形态分析
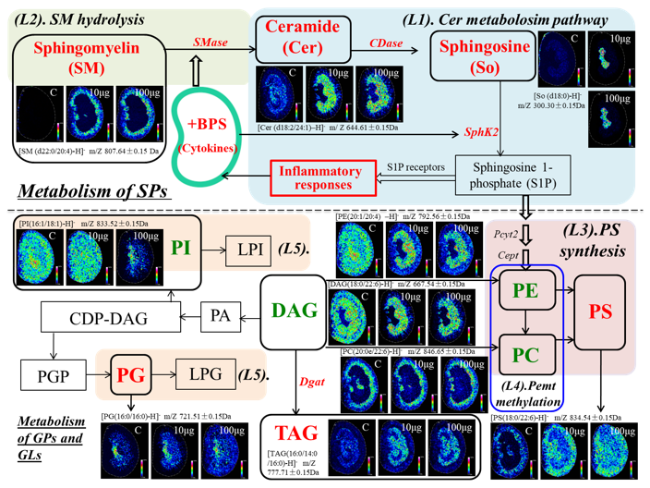
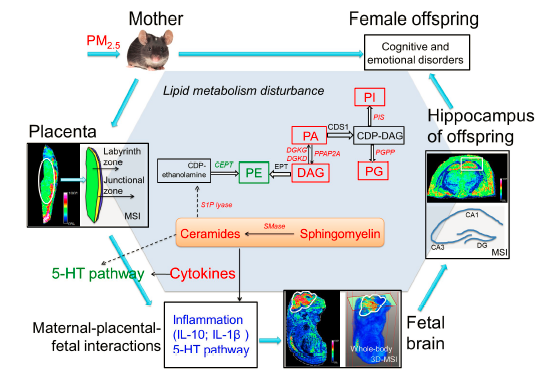
2.3 环境毒理的MSI和组学分析
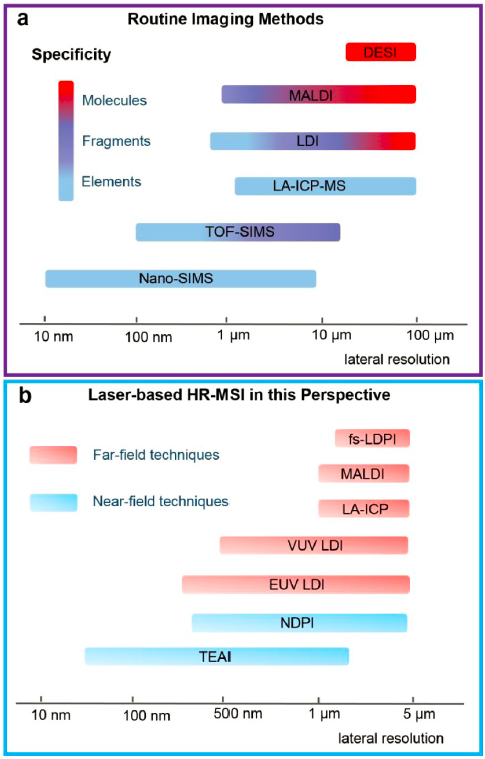
图3 MSI技术中空间分辨率的比较[28](a,常规方法;b,激光为基础的高分辨率成像方法。LDI,激光解吸电离;fs-LDPI,飞秒激光解吸和激光后电离;EUV,极紫外;NDPI,近场解吸后电离;TEAI,尖端增强消融电离)Fig.3 Comparison of spatial resolution in MSI[28](LDI, laser desorption ionization; fs-LDPI, fs laser desorption with laser post-ionization; EUV, extreme ultraviolet; NDPI, near-field desorption post-ionization; TEAI, tip-enhanced ablation and ionization) Copyright 2019, American Chemical Society |