Contents
1 引言
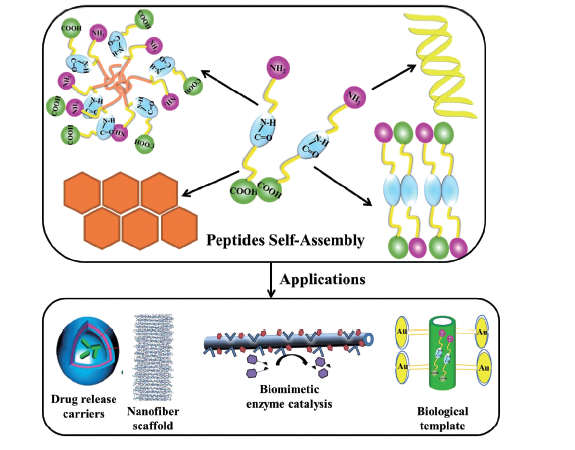
2 肽自组装机理
3 刺激响应型肽自组装研究进展
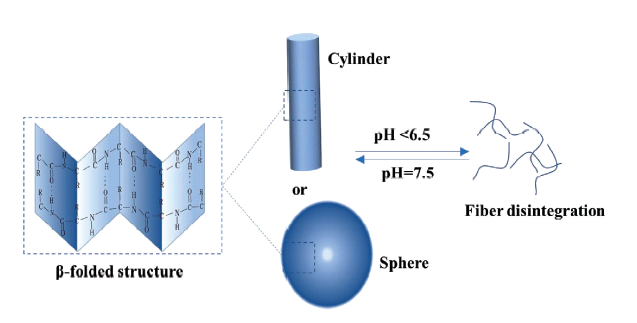
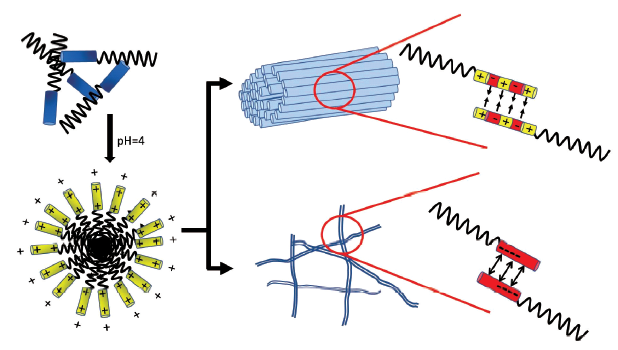
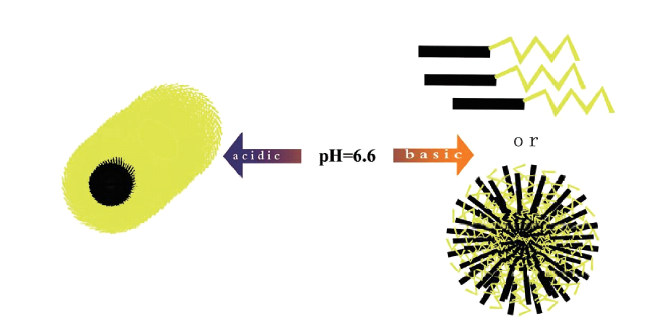
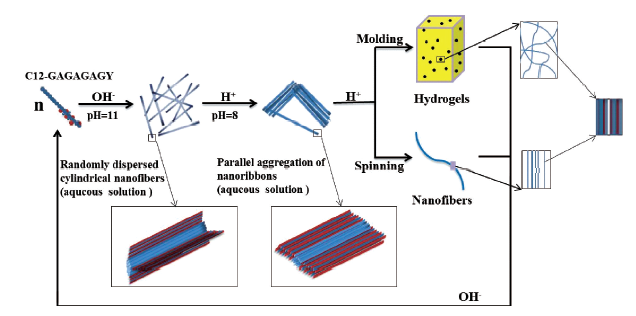
3.1 pH响应型肽自组装
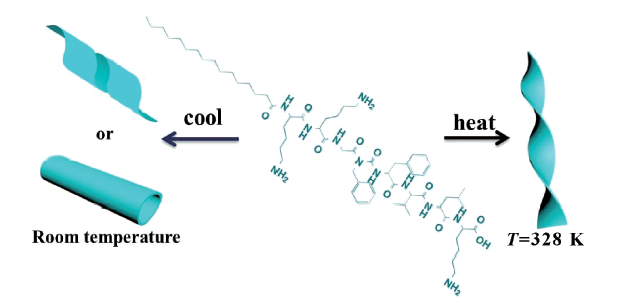
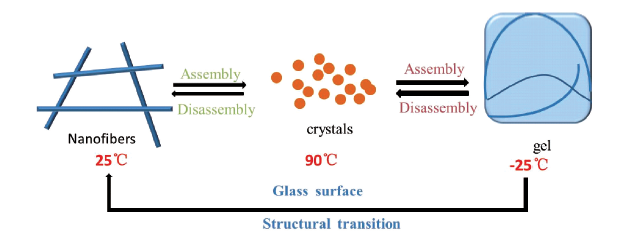
3.2 温度响应型肽自组装
3.3 溶剂响应型肽自组装
3.4 光响应型肽自组装
3.5 超声波响应型肽自组装
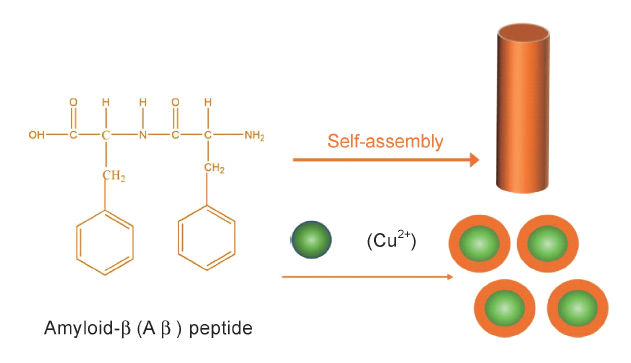
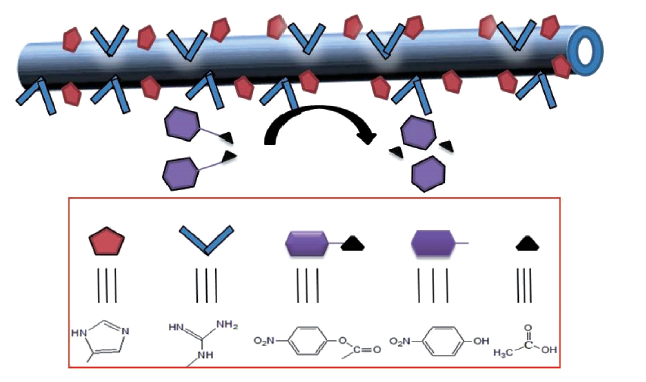
3.6 离子响应型肽自组装
4 肽自组装的应用
4.1 药物载体和释放
4.2 组织支架促进伤口愈合
4.3 脊髓修复
4.4 仿酶催化
4.5 制备光学材料
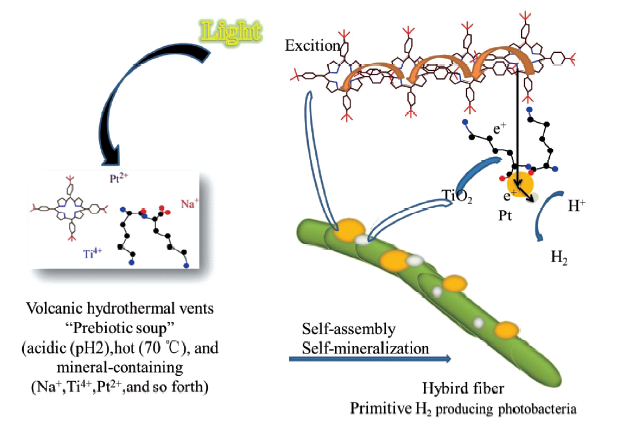
4.6 模拟光合成系统
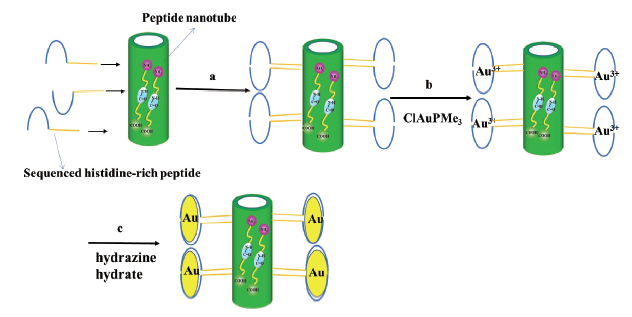
4. 生物模板
图15 制造金纳米线示意图(a)将含组氨酸序列的肽固定在模板的酰胺结合位点上(b)在测序组氨酸的肽上固定Au离子(c)Au纳米晶体生长[93]Fig.15 Scheme of the Au nanowire fabrication (a)Histidine sequence-containing peptides are immobilized on the amide binding site of the template (b) Au ion immobilization on the sequenced histidine-rich peptide and (c) Au nanocrystal growth[93] |